Brainbow
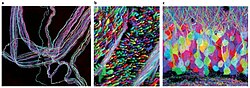
Brainbow är en teknik som har tagits fram för att enklare kunna identifiera och iaktta enskilda neuron i hjärnan. Med tekniken så kan individuella neuron urskiljas från närliggande med hjälp av fluorescerande proteiner. Då man slumpvis ger olika nivåer av fluorescerande protein (XFP), i antingen rött, blått eller grönt, till enskilda nervceller, så blir det möjligt att flagga varje enskild neuron med en egen färg. Det är denna stora färgvariation som har gett tekniken sitt namn, brainbow, som utgår ifrån engelskans namn för regnbåge, rainbow. Med ljusmikroskop kan man sedan följa de olika neuronen. [1][2] Tekniken presenterades för första gången i tidningen Nature 2007. Det var en forskargrupp vid Harvard, ledd av Jeff W. Lichtman och Joshua R. Sanes, båda professorer i molekylär och cellulär biologi, som tog fram denna teknik. Jean Livet, en del i forskargruppen sedan 2002, var den som kom på grundidén som tekniken bygger på. [3]
Sedan utvecklandet av Brainbow-tekniken 2007 har Brainbow bidragit till att utöka förståelsen för hjärnans konnektivitet. Tidigare histologiska tekniker tillåter endast kartläggning av ett fåtal neuron i taget. I jämförelse kan Brainbow-tekniken ge en tydlig bild av över 100 neuron i en och samma bild.
Utveckling
Brainbow utvecklades genom Lichtmans tidiga arbete på området. Redan i mitten av åttiotalet gjordes de första försöken att injicera möss med fluorescerande protein (XFP). Efter en elektrisk stimulering av axonen i det område som undersöktes injicerades XFP, som sedan togs upp på liknande sätt som en neurotransmittor av synapserna.
Senare utvecklade Lichtman genmodifierade möss, som i olika linjer bar på olika XFP-protein. Vissa av djuren i dessa linjer bar på olika starkt uttryckta XFP-protein, deras neuron var mer eller mindre fluorescerande. Genom att korsa dessa linjer fick man fram nya linjer som bar på fler varianter av XFP, två eller tre i samma neuron.
I takt med den tekniska utvecklingen på 2000-talet utvecklades ljusmikroskopin, och det blev möjligt att tydligare studera hjärnans nätverk. Lichtmans teknik kunde dock endast ge uttryck för ett fåtal färger i neuronen. För att få fram tydligare bild av de enskilda nervcellerna och deras samverkan behövdes det fler färger.
Som ett nästa steg, tog man fram multipla kopior av den genetiskt modifierade konstruktionen med XFP, och inympade dessa i genomet hos den specifika målarten. Detta resulterade i ett randomiserat uttryck av de olika XFP, vilket i sin tur fick cellerna att uppvisa en stor variation av färger. [1]
Metod

Brainbow utgår ifrån LoxP-Cre rekombineringssystem, i vilket enzymet Cre-rekombinas driver inversion eller excision av DNA mellan loxP-platser. Detta innebär att en del av sekvensen hamnar i bakvänd ordningsföljd (inversion) eller plockas bort helt (excision).[4][5] Man skapar alltså genetiska konstruktioner med flera XFP protein som flankeras av flera lox-platser. I sådana konstruktioner kan endast gener som direkt följer en promotor uttryckas. När denna konstruktion är skapad så aktiveras Cre-rekombinas. Denna inverterar eller exciserar DNA mellan ett par lox sekvenser. Det finns dock flera lox sekvenser som är inkompatibla med Cre (t.ex. loxP, loxN, lox2272). Om dessa sätts in i konstruktionen, är Cre-rekombinasen tvingad att välja mellan två inbördes exkluderande excisions möjligheter, vilket innebär att bara en möjlighet till återförvärv sker i en cell. Då valet av excisions/inversionsplats sker slumpmässigt i varje cell, kommer denna manipulation resultera i neurala celler märkta i olika nyanser. Ju fler olika XFP gener som används, desto fler färger kan potentiellt erhållas i nervcellen. [1]
Senare generationer av möss, som stammade från de möss som denna teknik först testats på, bar på flera olika kopior av dessa genetiska konstruktioner. Detta gjorde det möjligt för oberoende rekombinationer av de kombinerade XFP uttrycken, vilket resulterade i många nya färgkombinationer.
Detta beroende på att det vid tre transgena kopior är möjligt att erhålla tio olika färger. Tillägg av ytterligare kopior genererar i sin tur ett enormt antal nyanser - uppemot ett hundratal. [1]
Det finns idag tre typer av Brainbow-metoder. Originalmetoden innefattar både Brainbow-1 och Brainbow-2, där 1 är den ursprungliga metoden som beskrivits ovan och 2 den metod som upptäcktes vid studiet av senare generationer möss. Brainbow-3 är en senare metod som utvecklades 2013.
Brainbow-3 behåller Brainbow-1s loxP format, men använder sig av andra XFP-gener. Då dessa gener inte överlappar på samma sätt som de i Brainbow 1 och 2, och då de delar mindre av deras sekvenshomologi, gör detta att de lättare kan identifieras i immunhistokemiska protokoll. Brainbow-3 använder sig också av farnesylerade derivat av XFP, vilka ger en jämnare färgfyllning av nervcellerna.Med hjälp av fluorescerande mikroskopi, exempelvis med hjälp av konfokalmikroskop, så kan vi skapa färgglada bilder av olika områden i hjärnan. För att skapa dessa bilder av hela områden tas först tiotusentals bilder av ett område, för att sedan sammanställa dessa. När det är gjort kan man få fram ett komplett konnektomschema över ett visst område.[6]
Möss
Av de djur som har studerats med Brainbow är musens hjärna, med sina 75 miljoner neuron, den som mest påminner om vår egen. Det är också det första djuret som tekniken framgångsrikt testades på. Både Brainbow 1 och 2 utvecklades på möss.[3]
Användningsområden
Brainbow togs ursprungligen fram som ett komplement till, och som utveckling av, tidigare hjärnavbildningstekniker, såsom Golgimetoden och andra histologiska metoder. Dessa är dock problematiska att använda vid mer detaljerade kartläggningar av neuron och axon, bland annat eftersom de kan bara färga in nervceller i en begränsad färgskala, men framförallt då de ej kan användas in vitro. Med brainbow-tekniken kan en enskild nervcell märkas med upp till 100 olika fluorescerande nyanser, vilket gör att forskare kan identifiera och skilja mellan dendritiska och axonala processer. Det gör att nya slutsatser kan dras angående neuronala interaktioner och deras inverkan på beteende och funktion i hjärnan. Genom att jämföra skillnader i dessa processer kan man studera både neurologiska och psykologiska störningar.
Det ger också en helt ny möjlighet att bilda intrikata bilder av hjärnan, och gör att konnektiviteten kan kartläggas på ett helt nytt sätt. [1][3]
Begränsningar
Den största begränsningen som möter Brainbow, är det att framavlingen av möss med rätt uppsättning av XFP protein i sina nervceller innefattar både en komplex och tidskrävande process. Alla djur uppvisar inte heller en lika stark fluorescens, ett starkt uttryck hos föräldradjuret, innebär inte per automatik ett starkt uttryck hos avkomman.[3]
Ett annat problem som tidigt tillkom med de första metoderna var att färgerna i nervcellerna hade en tendens att blekna. Enligt Sanes, en av den ursprungliga forskargruppens ledare, kunde detta göra att man ”började med en brainbow, men slutade med en ’blue-bow’” då de andra färgerna hade försvunnit. Vid andra tillfällen var färgerna istället för ljusa, vilket ställde till problem vid användningen av ljusmikroskop, och ytterligare andra XPF protein fyllde inte helt ut cellerna. Många av dessa problem kunde dock avhjälpas i och med utvecklandet av Brainbow-3.[7]
Referenser
- ^ [a b c d e] Rojczyk-Gołębiewska, E., Pałasz, A., Worthington, J. J., Markowski, G., & Wiaderkiewicz, R.(2014). Neurolight - astonishing advances in brain imaging. The International Journal of Neuroscience, 125(2), 91–99. doi:10.3109/00207454.2014.914511.
- ^ ”Brainbow”. Arkiverad från originalet den 18 februari 2015. https://web.archive.org/web/20150218044748/http://cbs.fas.harvard.edu/science/connectome-project/brainbow. Läst 17 februari 2015.
- ^ [a b c d] Lichtman, J. W., Livet, J., & Sanes, J. R. (2008). A technicolour approach to the connectome. Nature Reviews. Neuroscience, 9, 417–422. doi:10.1038/nrn2391.
- ^ ”Inversion”. Arkiverad från originalet den 18 februari 2015. https://web.archive.org/web/20150218203156/http://genteknik.nu/typer-av-mutationer/. Läst 18 februari 2015.
- ^ ”Excision”. Arkiverad från originalet den 18 februari 2015. https://web.archive.org/web/20150218192958/http://mesh.kib.ki.se/swemesh/show.swemeshtree.cfm?mesh_eng=Dna+Repair. Läst 18 februari 2015.
- ^ Livet, J., Weissman, T. a, Kang, H., Draft, R. W., Lu, J., Bennis, R. a, … Lichtman, J. W. (2007). Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system. Nature, 450(November), 56–62. doi:10.1038/nature06293.
- ^ ”‘Brainbow,’ version 2.0”. http://news.harvard.edu/gazette/story/2013/05/brainbow-version-2-0/. Läst 17 februari 2015.
