Ewolucja biologiczna
Ewolucja biologiczna, ewolucja organiczna – zmiany cech całych grup organizmów następujące z biegiem pokoleń[1]. Procesy ewolucyjne powodują powstawanie bioróżnorodności na każdym poziomie organizacji biologicznej, w tym na poziomie gatunku, osobniczym i molekularnym[2]. W połowie XIX stulecia Charles Darwin sformułował teorię naukową ewolucji poprzez dobór naturalny, a odkrycie opublikował w monografii O powstawaniu gatunków (1859)[3].
Całe obecne życie na Ziemi pochodzi od wspólnego przodka[4][5][6]. Ostatni uniwersalny wspólny przodek, czyli ostatni wspólny przodek wszystkich dzisiejszych organizmów żywych, żył około 3,5–3,8 miliarda lat temu[7], aczkolwiek w 2015 opisano szczątki mogące stanowić pozostałości życia sprzed 4,1 miliarda lat w skałach Australii Zachodniej[8][9].
Powtarzające się powstawanie nowych gatunków (specjacja), zmiany w obrębie gatunków (anageneza) i wymieranie gatunków w obrębie historii życia na Ziemi demonstrowane są przez zespół wspólnych cech morfologicznych i biochemicznych, w tym sekwencje DNA[10]. Większe podobieństwo wspólnych cech obserwuje się u gatunków dzielących ze sobą późniejszego ostatniego wspólnego przodka, co może służyć rekonstrukcji biologicznego drzewa życia, bazującego na pokrewieństwie ewolucyjnym (filogenetyka), z wykorzystaniem zarówno gatunków współczesnych, jak i kopalnych. Zapis kopalny obrazuje progresję od wczesnego biogennego grafitu[11] przez maty mikrobowe[12][13][14] do skamieniałych organizmów wielokomórkowych. Istniejące wzorce bioróżnorodności wytworzyły się z udziałem specjacji i wymierań[15]. Powyżej 99% wszystkich gatunków, jakie kiedykolwiek żyły na Ziemi, wyginęło[16][17]. Obecnie Ziemię zamieszkują organizmy należące do 10–14 milionów gatunków[18], z których do tej pory opisano 1,2 miliona[19].
Ewolucja zachodząca poprzez dobór naturalny uwidacznia się w obserwacji, zgodnie z którą organizmy wydają na świat więcej potomstwa niż może przetrwać, w połączeniu z trzema faktami dotyczącymi populacji: 1) osobniki różnią się cechami morfologicznymi, fizjologicznymi i zachowaniem (wariancja fenotypowa), 2) różne cechy wiążą się z różną zdolnością do przetrwania i reprodukcji (różne dostosowanie), 3) cechy mogą być przekazywane z pokolenia na pokolenie (dziedziczność dostosowania)[3]. W kolejnych pokoleniach członkowie danej populacji zastępowani są przez potomstwo rodziców lepiej zaadaptowanych do przetrwania i rozrodu w danym środowisku, w którym zachodzi dobór naturalny. W tej teleonomii chodzi o tworzenie i zachowywanie cech dostosowanych do funkcji, które wypełniają[20]. Dobór naturalny stanowi jedyną znaną przyczynę powstawania adaptacji, ale nie jedyną przyczynę ewolucji. Inne, nieadaptacyjne procesy ewolucyjne obejmują mutacje, dryf genetyczny i przepływ genów na skutek migracji[21].
Na początku XX wieku powstała współczesna synteza ewolucyjna, integrująca klasyczną genetykę z darwinowską teorią ewolucji dzięki osiągnięciom genetyki populacyjnej. Wagę doboru naturalnego jako przyczyny ewolucji zaakceptowano w innych dziedzinach biologii. Co więcej, niegdyś uznawane poglądy na ewolucję, jak ortogeneza, XIX-wieczny ewolucjonizm czy inne poglądy dotyczące wrodzonego postępu w wielkoskalowych procesach ewolucyjnych przeszły do historii[22]. Naukowcy kontynuują badania różnych aspektów biologii ewolucyjnej przez formułowanie i testowanie hipotez, tworząc modele matematyczne biologii teoretycznej i teorie biologiczne, wykorzystując dane obserwacyjne i przeprowadzając doświadczenia w naturalnym środowisku i w laboratorium.
W sensie zastosowań praktycznych zrozumienie ewolucji wykorzystano na wielu polach nauki i przemysłu, włączając w to rolnictwo, medycynę człowieka i innych zwierząt oraz nauki o życiu w ogólności[23][24][25]. Odkrycia w biologii ewolucyjnej wywarły znaczny wpływ nie tylko na tradycyjne dziedziny biologii, ale również na inne dyscypliny akademickie, jak antropologia biologiczna czy psychologia ewolucyjna[26][27]. Obliczenia ewolucyjne, dział badań sztucznej inteligencji, stosuje zasady Darwina do rozwiązywania problemów informatycznych.
Historia myśli ewolucyjnej





Pomysł, jakoby organizmy jednego rodzaju mogły pochodzić od organizmów innego rodzaju, zaświtał jeszcze w filozofii przedsokratejskiej w starożytnej Grecji, u myślicieli takich, jak Anaksymander czy Empedokles[28]. Poglądy takie przetrwały do czasów rzymskich. Poeta i filozof Lukrecjusz poparł Empedoklesa w swym dziele De rerum natura (O naturze rzeczy)[29][30]. W przeciwieństwie do tych materialistycznych poglądów Arystoteles rozumiał wszystkie rzeczy materialne, nie tylko istoty żywe, jako niedoskonałe aktualizacje różnych ustalonych możliwości natury, co do których używał terminów forma, idea czy gatunek[31][32]. Wchodziło to w skład jego teleologicznego rozumienia natury, w której każda rzecz ma do odegrania swoją rolę w boskim, kosmicznym porządku. Modyfikacje tego poglądu stały się standardowym rozumieniem świata w średniowieczu, ulegając integracji z nauką chrześcijańską. Jednak Arystoteles nie żądał, by każdy realny typ organizmu żywego zawsze korespondował jeden do jednego z metafizyczną formą, dawał przykłady, jak mogły powstać nowe rodzaje istot żywych[33].
Niektórzy badacze uznają, że swoisty pierwowzór teorii Ewolucji powstał na przełomie IV i V wieku ne. i miał być dziełem świętego Aureliusza Augustyna z Hippony[34]. Święty ten uważał, powołując się na staro-łaciński przekład Mądrości Syracha, że Bóg stworzył wszystko jednocześnie, jednak nie wszystko pojawia się w tym samym czasie. Św. Augustyn zapożyczając stoicką doktrynę o „przyczynach zarodkowych”, stwierdził, że Bóg nie wszystko powołał do życia w gotowej postaci, ale w „zarodkach”, które rozwijają się, dopiero gdy powstają do tego dogodne warunki. Św. Augustyn jednak pod określeniem „przyczyny zarodkowe” nie miał na myśli biologicznych zarodków, a filozoficzne zasady określające finalny stan każdego z bytów[35].
W XVII wieku nowa metoda zastosowana w nauce wskrzesiła dociekania Arystotelesa. Szukano wyjaśnień naturalnych zjawisk w terminach praw fizycznych, takich samych dla wszystkich bytów widzialnych, a nie wymagających istnienia żadnych ustalonych naturalnych kategorii czy boskiego porządku kosmicznego. Jednak to nowe podejście powoli zakorzeniało się w naukach biologicznych, ostatnim bastionie idei ustalonych typów natury. John Ray zastosował jeden z wcześniejszych bardziej ogólnych terminów, gatunek, do typów zwierząt i roślin, jednak rygorystycznie identyfikował każdy typ organizmu żywego z gatunkiem i twierdził, że każdy gatunek można zdefiniować przez cechy utrwalające się z pokolenia na pokolenie[36][37].
Inni przyrodnicy tych czasów spekulowali nad ewolucyjnymi zmianami gatunków w czasie zgodnie z prawami natury. W 1751 Pierre Louis Maupertuis napisał o naturalnych modyfikacjach przebiegających podczas rozrodu oraz ich akumulacji na przestrzeni wielu pokoleń prowadzącej do powstania nowego gatunku[38]. Georges-Louis Leclerc zasugerował, że gatunki mogą degenerować w inne organizmy, a Erasmus Darwin zaproponował, że wszystkie zwierzęta ciepłokrwiste mogą pochodzić od pojedynczego mikroorganizmu (bądź „filamentu”)[39]. Pierwszy pełnoprawny schemat ewolucji wymyślił Jean-Baptiste Lamarck, tworząc „teorię transmutacji” w 1809[40]. Przewidywał on spontaniczne powstawanie prostych form życia, które potem rozwijały większą złożoność w równoległych liniach o nieodzownej tendencji do postępu, postulował, że na poziomie lokalnym linie te adaptują się do środowiska przez dziedziczenie zmian spowodowanych przez ich rodziców[41][42]. Ten ostatni proces nazwano później lamarkizmem[41][43][44][45]. Pomysły te zostały zdyskredytowane przez uznanych przyrodników jako spekulacje bez wsparcia empirycznego. W międzyczasie pomysły dobroczynnego projektu Raya zostały rozwinięte przez Paleya w Natural Theology or Evidences of the Existence and Attributes of the Deity (1802). Zaproponował on złożone adaptacje jako dowód na boski projekt, co spotkało się z podziwem Karola Darwina, w czasie gdy był on studentem Cambridge University[46][47][48][49].
Kluczowe zerwanie z poglądem o stałych klasach typologicznych bądź typach w biologii przyniosła teoria ewolucji poprzez dobór naturalny, sformułowana przez Karola Darwina w kategoriach zmiennych populacji. Na Darwina częściowo wpłynął An Essay on the Principle of Population z 1798 Thomasa Roberta Malthusa. Darwin zauważył, że wzrost populacji będzie prowadzić do walki o byt, w której korzystne warianty będą przeważały nad niekorzystnymi, które będą ginęły. W każdym pokoleniu wielu spośród potomstwa nie uda się przetrwać do wieku rozrodczego z powodu ograniczonych zasobów. To może wyjaśnić różnorodność roślin i zwierząt pochodzących od wspólnego przodka dzięki działaniu praw oddziaływających w ten sam sposób na wszystkie organizmy[50][51][52][53]. Darwin w 1832 rozpoczął obserwacje podczas trwającej 5 lat podróży na statku Beagle. W 1837 zaczął spisywać swe rozważania na temat przemiany gatunków. Swe wnioski zaprezentował w 1844 Lyellowi i Hookerowi. W 1858 otrzymał szkic pracy Alfreda Wallace’a. Praca tego badacza Archipelagu Malajskiego również opisywała dobór naturalny. Obie prace zostały przedstawione tego samego dnia, 1 lipca 1858, Towarzystwu Linneuszowskiemu w Londynie, co miało wyeliminować problem pierwszeństwa[54][55]. Pod koniec 1859 Darwin opublikował On the Origin of Species, wyjaśniając szczegółowo dobór naturalny w sposób, który zapewnił mu szeroką akceptację poglądu ewolucyjnego. Thomas Henry Huxley zastosował pogląd Darwina do człowieka. Wykorzystał paleontologię i anatomię porównawczą w dowodzeniu pochodzenia człowieka i małp człekokształtnych od wspólnego przodka. Niektórych myśl ta wzburzyła, gdyż przeczyła szczególnemu miejscu człowieka we Wszechświecie[56].
Dokładny mechanizm dziedziczności i pochodzenia nowych cech pozostawały zagadką. W związku z tym Darwin stworzył prowizoryczną hipotezę pangenezy[57]. W 1865 Gregor Mendel zauważył, że dziedziczenie cech przebiega w przewidywalny sposób, odkrywając niezależną segregację elementów dziedziczności (później nazwanych genami). Prawa Mendla ostatecznie wyparły darwinowską hipotezę pangenezy[58]. August Weismann dokonał ważnego rozróżnienia pomiędzy komórkami linii rozrodczej, z których powstają gamety (komórki jajowe i plemniki) oraz komórkami somatycznymi, budującymi ciało. Dziedziczność dotyczy jedynie komórek linii płciowych. Hugo de Vries połączył darwinowską pangenezę z weismannowskim rozróżnieniem komórek somatycznych i generatywnych oraz zaproponował, że pangeneza Darwina koncentruje się w jądrze komórkowym, a ulegając ekspresji, przenika do cytoplazmy, zmieniając budowę komórki. De Vries należał również do badaczy znających dobrze prace Mendla, uważających, że mendlowskie cechy korespondują z transferem dziedzicznym zmian w linii płciowej[59]. By wyjaśnić, jak powstają nowe warianty, Vries rozwinął teorię mutacji, prowadząc do czasowego pęknięcia między akceptującymi darwinowską ewolucję oraz biometrykami zgadzającymi się z de Vriesem[42][60][61]. W latach trzydziestych XX wieku pionierzy na polu genetyki populacyjnej, jak Ronald Fisher, Sewall Wright i John B.S. Haldane, oparli podstawy teorii ewolucji na statystyce. Fałszywy rozdźwięk pomiędzy teorią Darwina, mutacjami genetycznymi i dziedziczeniem mendlowskim został usunięty[62].
W drugiej i trzeciej dekadzie XX wieku synteza ewolucyjna połączyła dobór naturalny, mutacje i dziedziczenie mendlowskie w zunifikowaną teorię stosowaną w każdej dziedzinie biologii. Synteza ewolucyjna potrafiła wyjaśnić wzorce obserwowane w populacjach, w materiale kopalnym w paleontologii, a nawet złożone mechanizmy komórkowe biologii rozwoju[42][63]. Opisanie struktury DNA przez Watsona i Cricka w 1953 wyjaśniło fizyczny mechanizm dziedziczenia[64]. Biologia molekularna polepszyła rozumienie związków pomiędzy genotypem i fenotypem. Postęp nastąpił również w systematyce filogenetycznej. Obejmował on mapowanie przejść cech w porównywalnych i testowanych ramach i wykorzystanie drzew ewolucyjnych[65][66]. W 1973 biolog ewolucyjny Theodosius Dobzhansky napisał, że w biologii nic nie ma sensu, jeśli nie rozpatruje się tego w świetle ewolucji, ponieważ to ona rozświetliła relacje pomiędzy faktami z historii naturalnej uważanymi niegdyś za niełączące się ze sobą, umożliwiając stworzenie spójnej wiedzy pozwalającej na wyjaśnianie faktów i przewidywanie obserwacji dotyczących życia na Ziemi[67].
Od tamtych czasów synteza ewolucyjna była dalej poszerzana, by wyjaśnić zjawiska biologiczne na każdym poziomie organizacji biologicznej. To rozszerzenie zwane ewolucyjną biologią rozwoju, a nieformalnie „evo-devo”, skupia się na tym, jak zmiany między pokoleniami działają na wzorce zmian u pojedynczych organizmów (na rozwój)[68][69][70].
Próbując uwzględnić najnowsze rozważania ewolucjonistów, Blute w pracy z 2008 roku o definicji ewolucji stwierdził, że jej rozumienie jest obecnie tożsame z „mikroewolucją poprzez dobór naturalny”. Następnie stwierdził, iż najlepszą jej definicją jest: „każda zmiana w indukcyjnej kontroli rozwoju (morfologicznego, fizjologicznego lub behawioralnego) poprzez środowisko lub w konstrukcji środowiska poprzez rozwój, zmienia względną częstotliwość dziedzicznych elementów w populacji poza oczekiwane losowo wybrane warianty”[71].
Dziedziczność

Ewolucja organizmów żywych zachodzi poprzez zmiany w cechach podlegających dziedziczeniu (dziedzicznych). Przykładowo u ludzi kolor oczu podlega dziedziczeniu, osobnik może odziedziczyć na przykład brązowy kolor oczu po rodzicach[72]. Cechy dziedziczne kontrolowane są przez geny, a kompletny zestaw genów genomu (materiału genetycznego) danego organizmu nazywa się genotypem[73].
Zbiór obserwowalnych cech organizmu nazywa się z kolei jego fenotypem[74]. Cechy te biorą się z interakcji genotypu i wpływów środowiska[75]. W rezultacie wiele aspektów fenotypu organizmu nie jest dziedzicznych. Na przykład opalenizna skóry wynika z interakcji pomiędzy genotypem danej osoby a światłem, dlatego też osoba opalona nie przekaże tej cechy swemu potomstwu. Jednakże pewni ludzie opalają się łatwiej niż inni, co wynika z odmienności w genotypie. Radykalnym przykładem są osoby cierpiące na albinizm, które nie opalają się w ogóle i są niezwykle wrażliwe na oparzenia słoneczne[76].
Cechy dziedziczne przenoszone są z pokolenia na pokolenie poprzez DNA, cząsteczkę pozwalającą kodować informację genetyczną[73]. DNA jest długim biopolimerem budowanym przez cztery rodzaje zasad azotowych. Sekwencja zasad wzdłuż cząsteczki DNA odzwierciedla informację genetyczną, podobnie jak sekwencja liter odzwierciedla zdanie. Przed podziałem komórki DNA ulega kopiowaniu, w wyniku czego obie komórki potomne otrzymują wyjściową sekwencję DNA. Fragmenty cząsteczki DNA stanowiące jednostki funkcjonalne zwane są genami. Różne geny cechują się różnymi sekwencjami zasad azotowych. W komórce długie łańcuchy DNA tworzyć mogą skondensowane struktury określane mianem chromosomów. Specyficzne miejsce sekwencji DNA na chromosomie zwane jest locus. Jeśli sekwencja DNA w danym locus zmienia się pomiędzy osobnikami, różne jej formy nazywają się allelami. Sekwencja DNA może zmieniać się dzięki mutacjom, w efekcie czego powstają nowe allele. Jeśli zajdzie mutacja, nowy allel może wpływać inaczej na cechę kontrolowaną przez gen, co prowadzi do zmiany fenotypu organizmu[77]. Choć w niektórych przypadkach cecha odpowiada allelowi w prosty sposób, większość cech jest jednak bardziej złożona i kontrolowana przez wiele loci[78][79].
Niedawne badania potwierdziły ważne przykłady cech dziedzicznych niewyjaśnialnych za pomocą zmiany sekwencji nukleotydów w DNA. Zjawiska takie opisuje epigenetyka[80]. Metylacja DNA znacząca chromatynę, samopodtrzymujące się pętle metaboliczne, wyciszanie genów przez interferencję RNA oraz trójwymiarowe konformacje białek (takich jak priony) to obszary zachodzenia procesów epigenetycznych na poziomie organizmu[81][82]. Biolodzy rozwoju sugerują, że złożone interakcje sieci regulacyjnej genów i komunikacji międzykomórkowej mogą prowadzić do dziedzicznych zmian mogących odpowiadać za plastyczność rozwojową i kanalizację[83]. Dziedziczność może zachodzić nawet w większych skalach. Przykładowo dziedziczność ekologiczna poprzez proces budowy niszy ekologicznej definiuje się jako regularne i powtarzalne działania organizmów w środowisku. To generuje dziedziczenie efektów modyfikujących otoczenie i pętlę zwrotną dotykającą selekcji następnych pokoleń. Potomkowie dziedziczą geny plus charakterystykę środowiskową generowaną przez działanie ich przodków[84]. Inne przykłady dziedziczności niekontrolowanej bezpośrednio przez geny stanowią cechy kulturowe i symbiogeneza[85][86].
Zmienność


Fenotyp danego organizmu powstaje na skutek interakcji jego genotypu i wpływu środowiska życia. Za znaczną część zmienności fenotypowej odpowiada zmienność genetyczna[79], która w odróżnieniu od zmienności środowiskowej podlega dziedziczeniu. Synteza ewolucyjna definiuje zmiany ewolucyjne jako proces populacyjny, polegający na zmianie w czasie częstości osobników o różnych cechach, a więc i genotypach. Częstość poszczególnych alleli może rosnąć lub maleć względem innych form danego genu[74]. Wariacja znika, kiedy jeden z alleli osiąga fiksację, a więc kiedy albo znika z populacji, albo całkowicie zastępuje wszystkie inne allele[87].
Dobór naturalny doprowadzi do ewolucji tylko pod warunkiem istnienia wystarczającej zmienności genetycznej w populacji. Przed odkryciem genetyki mendlowskiej popularną hipotezą było mieszanie się cech. Oznaczałoby ono jednak drastyczny spadek zmienności genetycznej, czyniąc ewolucję nieprawdopodobną. Prawo Hardy’ego-Weinberga rozwiązuje ten problem, opisując, jak zmienność utrzymuje się w populacji przy założeniu dziedziczenia mendlowskiego. Częstości alleli w przypadku braku działania doboru naturalnego, zachodzenia mutacji, migracji i dryfu genetycznego pozostają stałe[88].
Zmienność tworzy się dzięki zachodzących w genomie mutacjom, tasowaniu się genów w trakcie rozmnażania płciowego oraz migracjom pomiędzy populacjami (przepływ genów). Pomimo stałego wprowadzania nowych wariantów i przepływu genów większość genomu jest identyczna u wszystkich przedstawicieli danego gatunku[89][90]. Jednak nawet niewielkie różnice w genotypie prowadzić mogą do dramatycznych zmian fenotypowych[potrzebny przypis], przykładowo szympansy i ludzie różnią się zaledwie w 5% swych genomów[91].
Mutacje

Mutacje to zmiany w sekwencji DNA. Zachodząca mutacja może zmienić produkt genu, uniemożliwiać funkcjonowanie genu, może też nie wywoływać żadnego efektu. Bazując na badaniach Drosophila melanogaster, zasugerowano, że jeśli mutacja zmienia łańcuch kodowanego przez gen białka, prawdopodobnie będzie powodować szkodliwy efekt. 70% takich mutacji wywołuje szkody, pozostałe są neutralne lub nieznacznie korzystne[92].
Mutacje obejmować mogą duplikacje dużych fragmentów chromosomów (zazwyczaj przez rekombinację genetyczną), wprowadzając dodatkowe kopie genów do genomu[93]. Dodatkowe kopie genów są głównym źródłem surowego materiału potrzebnego, by zachodziła ewolucja[94]. Jest to ważne, ponieważ większość nowych genów ewoluuje w obrębie rodzin genów z istniejących już genów, z którymi dzielą wspólnych przodków[95]. Na przykład ludzkie oko wykorzystuje 4 geny kodujące struktury światłoczułe, 3 w czopkach i jedną w pręcikach. Wszystkie one pochodzą od pojedynczego genu[96].
Nowe geny tworzyć się mogą z istniejących już genów, kiedy jedna z kopii zduplikowanego genu mutuje, zdobywając nową funkcję. Proces ten zachodzi łatwiej po duplikacji, gdyż zwiększa ona redundancję systemu. Jeden z genów może nabyć nową funkcję zamiast starej, którą wypełnia niezmutowany gen[97][98]. Inne rodzaje mutacji mogą nawet tworzyć całkowicie nowe geny, powstające z wcześniej niekodującego DNA[99][100].
Tworzenie się nowych genów może także wiązać się z duplikacją niewielkich odcinków kilku genów, które to fragmenty rekombinują, tworząc razem nową kombinację, pełniącą nową funkcję[101][102]. Kiedy nowe geny tworzą się z połączenia istniejących wcześniej odrębnie części, domeny działają jak moduły o prostych, niezależnych od siebie funkcjach. Moduły te można połączyć, tworząc nowe kombinacje o nowych, złożonych funkcjach[103]. Na przykład syntazy poliketydowe to duże enzymy tworzące antybiotyki. Zawierają do setki niezależnych domen, z których każda katalizuje jeden etap wieloetapowego procesu[104].
Płeć i rekombinacja
W przypadku organizmów rozmnażających się bezpłciowo geny dziedziczone są razem. Nie mieszają się w czasie rozrodu z genami innych organizmów. W przeciwieństwie do tej sytuacji potomstwo organizmów rozmnażających się płciowo dysponują losową mieszaniną chromosomów ich rodziców, rozdzielanych niezależnie. W pokrewnym procesie rekombinacji homologicznej organizmy płciowe wymieniają DNA pomiędzy dwoma odpowiadającymi sobie chromosomami[105]. Rekombinacja nie zmienia częstości alleli, zmienia natomiast grupowanie się ze sobą alleli różnych genów, dzięki czemu powstaje potomstwo o nowych kombinacjach alleli[106]. Płeć zwiększa zmienność genetyczną i może przyśpieszać tempo ewolucji[107][108].
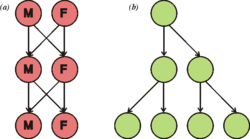
Dwukrotny koszt rozmnażania płciowego po raz pierwszy opisał John Maynard Smith[109]. Po pierwsze w przypadku istnienia płci tylko jedna z dwóch płci wydaje na świat młode. Nie dotyczy to gatunków hermafrodytycznych, jak większość roślin i wiele bezkręgowców. Po drugie organizm rozmnażający się płciowo przekazuje potomstwu jedynie połowę genów, a wartość ta zmniejsza się dalej z każdym następnym pokoleniem[110]. Dalej prowadzi to zazwyczaj do ewolucji dymorfizmu płciowego, pojawiającego się długo po wyewoluowaniu rozmnażania płciowego.[potrzebny przypis] Jednak rozmnażanie płciowe spotyka się często u eukariotów i organizmów wielokomórkowych. Hipoteza Czerwonej Królowej tłumaczy wagę rozmnażania płciowego, umożliwiającego ciągłą ewolucję i adaptację w odpowiedzi na koewolucję innych gatunków we wciąż zmieniających się środowisku[110][111][112][113].
Przepływ genów
Przepływ genów to ich wymiana pomiędzy populacjami, a nawet gatunkami[114]. Może stanowić źródło zmienności, nowej w danej populacji czy gatunku. Powoduje go przemieszczanie się osobników pomiędzy odrębnymi populacjami, na przykład dostanie się myszy z lądu na wyspę zamieszkaną przez inną ich populację czy też przemieszczenie się pyłku z trawy odpornej na metale ciężkie na trawę wrażliwą na ich obecność[115].
Przepływ genów pomiędzy gatunkami obejmuje tworzenie krzyżówek międzygatunkowych bądź horyzontalny transfer genów. To ostatnie pojęcie oznacza przeniesienie materiału genetycznego z jednego organizmu do innego, nie będącego potomstwem tego pierwszego. Najczęściej spotyka się go u bakterii[116]. Przyczynia się on do rozprzestrzeniania się oporności na antybiotyki – bakteria może zyskać gen oporności na antybiotyki poprzez szybki transfer od innego gatunku[117]. Zaobserwowano poziomy transfer genów bakteryjnych do eukariotów takich jak Saccharomyces cerevisiae i Callosobruchus chinensis[118][119]. Przykład transferu genów na większą skalę zapewniają Bdelloidea (podgromada Wrotków), posiadające geny dostarczone im przez bakterie, grzyby i rośliny[120]. DNA pomiędzy organizmami przenosić mogą także wirusy, potrafiące dokonywać transferu genów pomiędzy domenami[121].
Wielkoskalowy transfer genów zachodził także pomiędzy przodkami komórek eukariotycznych i bakteriami podczas tworzenia się chloroplastów i mitochondriów. Rekonstrukcja wczesnych etapów ewolucji na podstawie analizy materiału genetycznego głównych grup organizmów skłania do rezygnacji z przedstawiania zależności filogenetycznych w postaci drzewa i zastąpienie go przez pierścień życia. Eukarionty w takim ujęciu prawdopodobnie powstały w wyniku połączenia genomów proteobakterii i spokrewnionych z archeonami eocytów[122].
Mechanizmy

Z perspektywy neodarwinowskiej ewolucja zachodzi, gdy mają miejsce zmiany częstości alleli w populacji krzyżujących się między sobą organizmów[88]. Na przykład allele związane z ciemną barwą ciem stają się częstsze. Mechanizmy prowadzące do zmian w częstości alleli to dobór naturalny, dryf genetyczny, podwożenie, mutacje i przepływ genów.
Dobór naturalny
Ewolucja poprzez dobór naturalny to proces, w przebiegu którego cechy zwiększające zdolności organizmu do przeżycia lub rozrodu stają się częstsze w kolejnych pokoleniach w danej populacji. Mechanizm często uznaje się za oczywisty, ponieważ wynika on analitycznie z trzech faktów[3]:
- w obrębie populacji istnieje zmienność dotycząca morfologii, fizjologii i zachowania (zmienność fenotypowa)
- różne cechy wiążą się z różną zdolnością do przeżycia i reprodukcji (różne dostosowanie)
- cechy te mogą być przekazywane z pokolenia na pokolenie (dziedziczność dostosowania).
Organizmy wydają na świat więcej potomstwa, niż może przeżyć, co powoduje konkurencję o przetrwanie i możliwość rozrodu. W efekcie osobniki o cechach przynoszących im większe korzyści względem ich konkurentów z większym prawdopodobieństwem przekażą swe cechy następnemu pokoleniu, niż organizmy posiadające cechy nie gwarantujące takiej przewagi[123].
Centralnym pojęciem związanym z doborem naturalnym jest dostosowanie organizmu[124]. Oznacza ono zdolność osobnika do przetrwania i reprodukcji, które determinują wkład genów tego organizmu w następnym pokoleniu[124]. Dostosowanie nie jest jednak tożsame z całkowitą liczbą potomstwa, wskazuje raczej proporcję organizmów noszących geny danego osobnika w następnych pokoleniach[125]. Przykładowo jeśli pewien organizm ma duże zdolności przetrwania i szybko się rozmnaża, ale jego potomstwo jest zbyt małe i słabe, by przetrwać, będzie miał on nieznaczny wkład genów w przyszłych pokoleniach i jego dostosowanie będzie niewielkie[124].
Jeśli allel zwiększa dostosowanie w stopniu większym, niż inne allele tego samego genu, wtedy z każdym kolejnym pokoleniem będzie on występował w danej populacji częściej. Związana z nim cecha ulega pozytywnemu doborowi naturalnemu. Jako przykład wymienia się cechy zwiększające szanse przeżycia i zwiększające rozrodczość. Odwrotnie – mniejsze dostosowanie spowodowane posiadaniem allela mniej korzystnego czy wręcz szkodliwego skutkuje jego coraz rzadszym pojawianiem się w następnych pokoleniach, cecha będzie usuwana z populacji przez dobór[126]. Co ważne, dostosowanie danego allela nie jest jego ustaloną cechą. Jeśli zmienią się warunki środowiskowe, cecha wcześniej neutralna bądź szkodliwa może okazać się korzystna, a cecha niegdyś korzystna może stać się szkodliwa[77]. Jednak nawet jeśli kierunek działania doboru naturalnego może odwrócić się w ten sposób, cecha wyeliminowana z populacji w przeszłości nie może powtórnie wyewoluować w identycznej formie, co opisuje prawo Dollo[127][128].
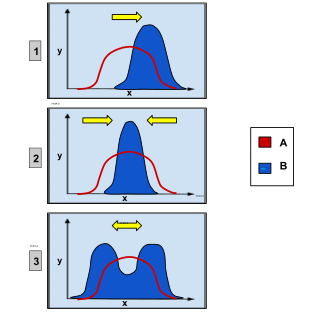
· 1) dobór kierunkowy, który faworyzuje pojedynczy fenotyp skrajny
· 2) dobór stabilizujący, faworyzujący fenotyp pośredni
· 3) dobór różnicujący, czyli rozrywający, który faworyzuje oba fenotypy skrajne
Dobór naturalny w populacji dotyczący cechy mogącej przyjąć przedział wartości, jak wysokość, można skategoryzować, wyróżniając 3 jego rodzaje. Po pierwsze dobór kierunkowy zmieniający w czasie średnią wartość rzeczonej cechy, na przykład organizmy stają się coraz wyższe[129]. Po drugie dobór rozrywający zwany też różnicującym oznacza dobór skrajnych wartości cechy i często skutkuje dwiema wartościami cechy częstszymi od pozostałych, natomiast wartość pośrednia jest eliminowana z populacji. Byłoby tak, gdyby organizmy wysokie lub niskie osiągały przewagę nad tymi średniego wzrostu. Istnieje w końcu dobór stabilizujący, w którym dobór działa przeciwko skrajnym wartościom rozpatrywanej cechy. Dobór ten zmniejsza zmienność cechy wokoło jej średniej wartości[123][130]. Dobór taki spowodowałby przykładowo podobną wysokość wszystkich organizmów.
Specjalnym przypadkiem doboru naturalnego jest dobór płciowy, w przypadku którego doborowi podlega cecha zwiększająca sukces rozrodczy poprzez zwiększanie atrakcyjności organizmu dla potencjalnych partnerów rozrodczych[131]. Cechy, które wyewoluowały dzięki doborowi płciowemu, są szczególnie dobrze wyrażone u samców pewnych gatunków zwierząt. Choć faworyzowane przez dobór płciowy, cechy takie jak niewygodne poroże, nawoływania samców, duże rozmiary ciała czy jaskrawe barwy często przyciągają uwagę drapieżników, co redukuje przeżywalność tych samców[132][133]. Ta wada równoważona jest przez większy sukces reprodukcyjny samców prezentujących takie w widoczny sposób upośledzające cechy[134].
Dobór naturalny najogólniej czyni naturę miarą prawdopodobieństwa przetrwania osobników bądź ich cech. Słowo natura odnosi się tutaj do ekosystemu – systemu, w którym organizmy oddziałują ze wszystkimi jego elementami, fizycznymi lub biologicznymi, w swym lokalnym środowisku. Eugene Odum, twórca ekologii, zdefiniował ekosystem jako każdą jednostkę obejmującą wszystkie organizmy na danym obszarze oddziałujące ze środowiskiem fizycznym, w związku z czym energia przepływa przez jasno zdefiniowaną strukturę troficzną, zróżnicowanie biotyczne i cykle substancji (np. wymiana między materią ożywioną a nieożywioną) w obrębie systemu[135]. Każda populacja w ekosystemie zajmuje odrębną niszę ekologiczną, własną pozycję o odrębnych powiązaniach z innymi częściami systemu. Zależności te wiążą się z przebiegiem życia organizmu, jego pozycją w łańcuchach pokarmowych i jego zasięgiem geograficznym. To szerokie rozumienie natury umożliwia naukowcom nakreślenie specyficznych oddziaływań, które wspólnie składają się na dobór naturalny.
Dobór naturalny działa na różnych poziomach, jak geny, komórki, organizmy, grupy organizmów, gatunki[136][137][138]. Może też jednocześnie działać na wielu poziomach[139]. Przykładowo dobór zachodzący na poziomie niższym od poziomu organizmu dotyczy genów zwanych transpozonami, zdolnych do replikacji i rozprzestrzeniania się w obrębie genomu[140]. Dobór na poziomie wyższym niż osobniczy występuje w przypadku doboru grupowego i może doprowadzić do ewolucji współpracy[141].
Mutacje tendencyjne
Będąc głównym źródłem zmienności, mutacje działają również w innym mechanizmie ewolucyjnym.[potrzebny przypis] Kiedy na poziomie molekularnym istnieją różne prawdopodobieństwa zajścia różnych mutacji, mówi się o mutacjach tendencyjnych (mutation bias)[142]. Jeśli dwa genotypy, niech przykładowo jeden zawiera w pewnym miejscu nukleotyd G, a drugi nukleotyd A, cechują się jednakowym dostosowaniem, ale mutacja zmieniająca G na A zdarza się częściej, niż zmieniająca A na G, ewolucja będzie przebiegała w kierunku A[143]. Różnice w mutacjach tendencyjnych insercja-delecja w różnych taksonach mogą prowadzić do ewolucji odmiennych wielkości genomów[144][145]. Zjawisko takie obserwowano także w ewolucji morfologii[146][147]. Przykładowo mogą w końcu prowadzić do genetycznej asymilacji cechy wcześniej powstającej na skutek oddziaływań środowiskowych[148][149].
Opisany efekt nakłada się na inne procesy. Jeśli dobór będzie faworyzował jedną z dwu możliwych mutacji, a nie będzie żadnej dodatkowej korzyści z posiadania obydwu, wtedy mutacja pojawiająca się częściej będzie tą, która ulegnie utrwaleniu z największym prawdopodobieństwem[150][151]. Mutacje prowadzące do utraty funkcji genu są znacznie częstsze niż mutacje prowadzące do wytworzenia się nowego, w pełni funkcjonalnego genu. Większość mutacji utraty funkcji podlega więc negatywnej selekcji. Kiedy jednak dobór ten działa słabo, efekt mutacji tendencyjnej w kierunku utraty funkcji może wpływać na proces ewolucji[152]. Na przykład zwierzę przystosowujące się do życia w jaskiniach nie potrzebuje już pigmentów, są one więc tracone[153]. Taki rodzaj utraty funkcji może zachodzić dzięki mutacjom tendencyjnym lub z powodu kosztów, które pociąga za sobą każda funkcja. Kiedy więc znika przynoszony przez daną funkcję zysk, dobór naturalny prowadzi do jej usunięcia. Utrata zdolności sporulacji Bacillus subtilis w ewolucji laboratoryjnej wydaje się wynikać raczej właśnie z mutacji tendencyjnych, a nie z doboru naturalnego przeciwko kosztom utrzymywania zdolności wytwarzania spor[154]. Kiedy nie występuje dobór faworyzujący utratę funkcji, szybkość ewolucji jej utraty zależy bardziej od tempa zachodzenia mutacji, niż od efektywnej wielkości ewolucji[155]. Wskazuje to, że proces napędzany jest bardziej mutacjami tendencyjnymi, niż dryfem genetycznym.
Dryf genetyczny
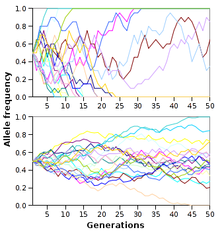
Dryf genetyczny polega na zmianie częstości alleli z jednego pokolenia na następne z powodu działania błędu próby[156]. W rezultacie kiedy selekcja jest względnie słaba bądź też w ogóle nie zachodzi, częstości alleli losowo rosną bądź maleją (zachodzi błądzenie losowe). Dryf zatrzymuje się, dopiero gdy allel ulegnie fiksacji: albo zostanie wyeliminowany z populacji, albo zastąpi w niej wszystkie inne allele. Dryf genetyczny może w ten sposób wyeliminować z populacji niektóre allele przez przypadek. Nawet w przypadku niezachodzenia doboru naturalnego dryf genetyczny może doprowadzić do powstania dwóch oddzielnych populacji, które początkowo miały taką samą strukturę genetyczną, ale uległy dryfowi w dwóch odmiennych kierunkach, osiągając przez to odmienne zbiory alleli[157].
Zmierzenie względnej wagi doboru i procesów neutralnych, do których zalicza się dryf, zazwyczaj przedstawia trudności[158]. Porównywane znaczenie procesów adaptacyjnych i nieadaptacyjnych dla zachodzenia zmian ewolucyjnych stanowi pole działań współczesnych badań[159].
W teorii neutralnej ewolucji molekularnej większość zmian ewolucyjnych stanowi efekt fiksacji mutacji neutralnych przez dryf genetyczny[160]. Stąd w tym modelu większość zmian genetycznych w populacji wynika ze stałej presji mutacyjnej i dryfu[161]. Taka forma teorii neutralnej została obecnie w dużym stopniu zarzucona, jako że nie zgadza się ze zmiennością genetyczną obserwowaną w naturze[162][163]. Jednak istnieje współcześniejsza i lepiej wsparta wersja tego modelu, prawie neutralna teoria ewolucji molekularnej, w której mutacje efektywnie neutralne w niewielkiej populacji niekoniecznie muszą być neutralne w populacji liczebniejszej[123]. Inne alternatywne teorie proponują, że wpływ dryfu genetycznego redukują inne stochastyczne procesy ewolucyjne, jak podwożenie[156][164][165].
Czas, w którym allel neutralny ulegnie utrwaleniu przez dryf genetyczny, zależy od liczebności tej populacji, fiksacja zachodzi szybciej w niewielkich populacjach[166]. Liczba osobników w populacji nie jest krytyczna, znacznie ważniejsza jest efektywna wielkość populacji[167]. Zazwyczaj ustępuje ona liczebności populacji, ponieważ wpływają nań czynniki takie, jak poziom chowu wsobnego i etap cyklu życiowego, w którym populacja jest najmniejsza[167]. Efektywna wielkość populacji nie musi być jednakowa dla każdego genu w danej populacji[168].
Podwożenie
Rekombinacja umożliwia allelom leżącym na tej samej nici DNA na niezależny rozdział. Jednak rekombinacja nie zachodzi często, średnio dwukrotnie na chromosom na pokolenie. W efekcie geny umiejscowione blisko siebie na chromosomie nie zawsze będą segregować niezależnie, zamiast tego będą raczej dziedziczone razem, co określa się mianem sprzężenia[169]. Tę tendencję mierzy się poprzez sprawdzenie, jak często 2 allele występują razem na jednym chromosomie w porównaniu z przewidywaniami – mówi się tutaj o nierównowadze sprzężeń. Zbiór alleli zazwyczaj dziedziczony razem zwany jest haplotypem. Istotne jest, kiedy jeden allel danego haplotypu daje silną przewagę selekcyjną. Dobór naturalny powoduje wtedy zjawisko zwane selective sweep – inne allele tego haplotypu również stają się częstsze w populacji. Efekt ten nazywa się podwożeniem[170]. Podwożenie spowodowane faktem genetycznego powiązania genów neutralnych z podlegającymi doborowi może być częściowo ograniczone przez efektywną wielkość populacji[164].
Przepływ genów
Przepływ genów polega na wymianie genów między populacjami lub gatunkami[114]. Może zachodzić dzięki przemieszczającym się osobnikom czy też gametom, pod warunkiem, że biorą udział w rozrodzie po dotarciu do nowej populacji. Proces ten można opisać modelem traktującym populacje jako odrębne lub też modelem izolacji na odległość, w którym każdemu osobnikowi przypisuje się sąsiedztwo i im większy dystans pomiędzy dwoma osobnikami, tym mniejsze prawdopodobieństwo wspólnego rozrodu. W takim ujęciu populację tworzą nakładające się na siebie sąsiedztwa. Modele przepływu genów można też podzielić na zakładające stałe tempo przepływu genów oraz opisujące sytuację, w której dana populacja wymiera w niektórych obszarach jej występowania, gdzie zastępują ją osobniki z innych populacji[171].
Podczas tworzenia syntezy ewolucyjnej Sewall Wright rozwinął swą teorię zmiennej równowagi, uznającą przepływ genów między częściowo izolowanymi populacjami za ważny aspekt ewolucji adaptacyjnej[172]. Jednak obecnie znaczenie tej teorii jest krytykowane, jako że nie uwzględnia ona pewnych ograniczeń przepływu genów oraz z powodu pewnych trudności związanych ze złożonymi adaptacjami[173].
Skutki
Ewolucja wpływa na każdy aspekt budowy i zachowania organizmów. Najbardziej rzucają się w oczy swoiste adaptacje powstałe na skutek działania doboru naturalnego. Zwiększają one dostosowanie, wspomagając takie czynności, jak żerowanie, unikanie drapieżników czy przyciąganie partnera. Dobór może też doprowadzić do rozwoju współpracy między osobnikami, czy to poprzez wspieranie własnych krewnych, czy też symbiozę. W dłuższym czasie ewolucja tworzy nowe gatunki, rozdzielając populacje na grupy, które dzieli bariera uniemożliwiająca rozród.
Skutki działania ewolucji dzieli się, biorąc pod uwagę czas jej działania, wyróżniając makroewolucję i mikroewolucję. Makroewolucja odbywa się na poziomie wyższym od gatunku[174], obejmuje specjację i wymieranie. Mikroewolucja obejmuje natomiast mniejsze zmiany ewolucyjne w obrębie gatunku czy pojedynczej populacji, w szczególności zaś zmiany częstości poszczególnych alleli i adaptacje[175]. Ogólnie skutki makroewolucji pojawiają się po dłuższym czasie zachodzenia mikroewolucji[176]. W związku z tym podział na makro- i mikroewolucję nie jest fundamentalny, różnica dotyczy tylko czasu potrzebnego dla zajścia zmian[177]. Jednak w przypadku makroewolucji ważna może być cecha całego gatunku. Na przykład duża zmienność między osobnikami pozwala gatunkowi szybko adaptować się do nowego siedliska, zmniejszając prawdopodobieństwo wyginięcia, podczas gdy szeroki zasięg geograficzny zwiększa szanse na specjację, podwyższając prawdopodobieństwo izolacji części populacji. W tym sensie mikro- i makroewolucja mogą pociągać za sobą dobór na odmiennych poziomach: w przypadku mikroewolucji chodzi o poziom genów i organizmów, w makroewolucji chodzi o dobór na poziomie gatunku, związany z tempem specjacji czy wymierania[178][179][180].
Choć wyewoluowały gatunki cechujące się wysoką złożonością, istnieją one jako efekt uboczny wielości organizmów, a proste formy życia ciągle pozostają najczęstszymi w biosferze[181]. Na przykład przytłaczająca większość organizmów stanowią mikroskopowe prokarioty, stanowiące pomimo niewielkich rozmiarów około połowy światowej biomasy[182] i przyczyniają się do znaczącej większości ziemskiej bioróżnorodności[183]. Proste organizmy żywe dominują na Ziemi przez całą historię istnienia życia i do dziś stanowią główną formę życia. Złożone formy życia wydają się bardziej zróżnicowane tylko dlatego, że są bardziej zauważalne[184]. W związku z tym ewolucja mikroorganizmów jest szczególnie ważna w dzisiejszej biologii ewolucyjnej, tym bardziej że ich szybki rozród pozwala na eksperymentalne badania ewolucji oraz obserwacje ewolucji i tworzenia adaptacji w czasie rzeczywistym[185][186].
Adaptacje
Adaptacja to proces, w którym organizmy lepiej przystosowują się do siedlisk. Jak pisze Mayr[187], nie jest to trwały stan, ale produkt kreatywnej przeszłości i zarazem trwający, dynamiczny proces. Oxford Dictionary of Science definiuje adaptację jako każdą zmianę w strukturze lub funkcjonowaniu kolejnych pokoleń, które czynią je lepiej przystosowanymi do środowiska[188]. Termin ten może odnosić się również do cechy ważnej dla przetrwania organizmu, na przykład adaptacja zębów konia do żywienia się trawą. Poprzez użycie słowa „adaptacja” na określenie procesu ewolucyjnego oraz „cecha adaptacyjna” na oznaczenie jej produktu (części ciała lub funkcji) rozróżnić można oba znaczenia. Adaptacja jest efektem doboru naturalnego[189]. Theodosius Dobzhansky wyróżnia następujące pojęcia:
- Adaptacja – proces ewolucyjny, w którym organizm staje się lepiej przystosowany do życia w jego siedlisku bądź siedliskach[190]
- Adaptacyjność – stan bycia zaadaptowanym, stopień, w jakim organizm jest zdolny do życia i rozrodu w danym zestawie siedlisk[191]
- Cecha adaptacyjna – aspekt wzorca rozwojowego organizmu pozwalający mu zwiększyć prawdopodobieństwo przetrwania bądź reprodukcji[192].
Adaptacją może być zarówno wytworzenie się nowej cechy, jak też utrata cechy ancestralnej. Przykłady obu widać w bakteryjnej adaptacji do antybiotyków. Zmiany genetyczne wytwarzające oporność na antybiotyki polegać mogą na modyfikacji targetu molekularnego antybiotyku bądź na zwiększeniu aktywności transportera wypompowującego lek z komórki[193]. Za zadziwiający przykład uchodzi wyewoluowanie przez Escherichia coli zdolności do wykorzystywania kwasu cytrynowego jako składnika odżywczego w długoterminowym eksperymencie laboratoryjnym[194] czy też wytworzenie przez Flavobacterium nowego enzymu pozwalającego tej bakterii rosnąć na odpadach z produkcji nylonu[195][196], jak też wyewoluowanie u bakterii glebowej Sphingobium całkowicie nowego szlaku metabolicznego degradującego syntetyczny pestycyd pentachlorofenol[197][198]. Interesujący, ale wciąż kontrowersyjny pomysł zakłada, że niektóre adaptacje mogą zwiększać zdolność organizmów do generowania różnorodności genetycznej i adaptacji przez dobór naturalny (zwiększająca się ewoluowalność)[199][200][201][202][203].
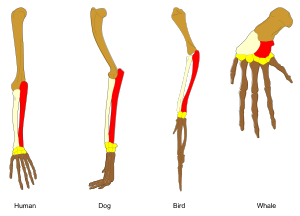
Adaptacje powstają poprzez stopniowe modyfikacje istniejących struktur. W efekcie struktury o podobnej organizacji wewnętrznej mogą nabywać odmienne funkcje u spokrewnionych ze sobą organizmów. Pojedyncza cecha ancestralna ulega przystosowaniu do pełnienia różnych funkcji w odmienny sposób. Przykładowo kości kończyny górnej nietoperzy posiadających skrzydła są bardzo podobne do odpowiadających im kości łapy myszy czy posiadających dłonie naczelnych. Wszystkie te kończyny wywodzą się z tej samej struktury obecnej u ich wspólnego przodka[205]. Jednakże jako że wszystkie organizmy żywe są ze sobą spokrewnione[206], nawet organizmy wydające się dzielić ze sobą niewielkie podobieństwa budowy bądź też nie dzielić żadnych, jak w przypadku oczu stawonogów, mięczaków i kręgowców czy też kończyn i skrzydeł stawonogów i kręgowców, mają narządy wykorzystujące zbiór homologicznych genów kontrolujących ich powstawanie i działanie. Zjawisko to nazywa się głęboką homologią[207][208].
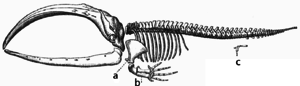
W trakcie ewolucji pewne struktury mogą tracić swe pierwotne funkcje. Tworzą się w ten sposób struktury szczątkowe[209]. Pewne struktury mogą pełnić nieznaczące funkcje bądź w ogóle nie posiadać żadnych u współczesnych gatunków, jednakże wypełniać czytelne role u przodków tych organizmów bądź u gatunków blisko spokrewnionych. Można tu wymienić pseudogeny[210], niedziałające pozostałości oka u ślepych ryb jaskiniowych[211], skrzydła nielotnych owadów[212], obecność kości udowych u waleni i węży[204] oraz cechy płciowe u organizmów rozmnażających się bezpłciowo[213]. U człowieka przykłady stanowią zęby mądrości[214], kość guziczna[209], wyrostek robaczkowy[209], ale też cechy behawioralne, jak gęsia skórka[215][216] i odruchy pierwotne[217][218][219].
Jednakże wiele cech, które zdają się być prostymi adaptacjami, to w rzeczywistości egzaptacje. Struktura pierwotnie przystosowana do pełnienia jakiejś funkcji może przypadkowo okazać się pomocna w wypełnianiu innej funkcji[220]. Przykładowo afrykańska jaszczurka tęczowa wykształciła niezwykle płaską głowę, co przydaje się podczas ukrywania się w szczelinach. Można to zaobserwować również u jej najbliższych krewnych. Jednak jej głowa jest aż tak płaska, że pomaga w locie szybowym z drzewa na drzewo[220]. W obrębie komórki struktury molekularne takie jak wić bakteryjna[221] i mechanizm sortowania białek[222] wyewoluowały dzięki zaprzęgnięciu istniejących przed ich powstaniem białek wykonujących wtedy inne funkcje[175]. Inny przykład stanowi wykorzystanie enzymów glikolizy czy utylizacji ksenobiotyków jako białek strukturalnych – w ten sposób powstały krystaliny soczewki oka[223][224].
Obszar współczesnych badań biologii rozwoju obejmuje rozwojową podstawę adaptacji i egzaptacji[225]. Badania te obejmują powstanie i ewolucję rozwoju zarodkowego oraz jak modyfikacje rozwoju i procesów rozwojowych prowadzą do powstawania nowych cech[226][227]. Badania te wykazały, że ewolucja może zmieniać rozwój, tworząc nowe struktury takie jak kości zarodkowe rozwijające się w kości żuchwy u jednych zwierząt, a w kosteczki słuchowe u innych[228]. Możliwe jest również, że struktura tracona w trakcie ewolucji pojawia się ponownie w wyniku zmian genów kierujących rozwojem, na przykład mutacja powodująca w rozwijającym się zarodku kurczaka rozwój zębów przypominających spotykane u krokodyli[229]. Obecnie staje się jasne, że większość zmian w formie organizmów spowodowana jest modyfikacjami niewielkiego zbioru konserwatywnych genów[230].
Koewolucja

Interakcje pomiędzy organizmami mogą prowadzić zarówno do konfliktu, jak i do współpracy. W przypadku interakcji pomiędzy dwoma gatunkami, jak patogen i jego żywiciel bądź drapieżnik i ofiara, gatunki mogą wytwarzać powiązane zestawy adaptacji. Ewolucja jednego gatunku powoduje w takim układzie adaptację drugiego. I odwrotnie: zmiany zachodzące w drugim gatunku wywołują powstawanie nowych cech adaptacyjnych u pierwszego. Taki cykl doboru naturalnego i odpowiedzi nań nazywany jest koewolucją[231]. Przykładem jest produkcja tetradotoksyny przez pacyfotrytona szorstkiego oraz ewolucja oporności na tę toksynę przez polującego na rzeczonego płaza drapieżnika, pończosznika prążkowanego. W tej parze drapieżnik-ofiara ewolucyjny wyścig zbrojeń doprowadził do wysokich stężeń tetradotoksyny u traszki i odpowiadającej mu wysokiej oporności na toksynę u węża[232].
Kooperacja
Jednak nie zawsze koewolucja musi prowadzić do konfliktu[233][234]. Na drodze ewolucji powstało również wiele interakcji korzystnych dla obu uczestników. Przykładowo ekstremalny poziom współpracy osiągnęły rośliny i grzyby zaangażowane w mikoryzę. Grzyby rosną na ich korzeniach i wspomagają roślinę, absorbując substancje odżywcze z gleby[235]. Roślina odwzajemnia się, zaopatrując grzyby w węglowodany, które wytwarza drogą fotosyntezy. Grzyby rosną właściwie w komórkach roślin, pozwalających im na wymianę substancji odżywczych z gospodarzem dzięki hamowaniu jego układu odpornościowego[236].
Koalicje wiążące organizmy tego samego gatunku również powstały drogą ewolucji. Ekstremalny przypadek stanowi tym razem eusocjalność, spotykana u owadów społecznych, jak Apiformes, termity, mrówkowate. Niepłodne osobniki karmią w ich populacjach i chronią niewielką liczbę członków kolonii zdolnych do rozrodu. W mniejszej skali komórki somatyczne budujące ciało zwierzęcia ograniczają samopowielanie, utrzymując w ten sposób stabilność organizmu, utrzymującego z kolei niewielką liczbę komórek rozrodczych, odpowiedzialnych za powoływanie na świat potomstwa. Komórki somatyczne odpowiadają na specyficzne sygnały informujące je, czy rosnąć, pozostawać w stanie niezmienionym, czy umierać. Jeśli komórka ignoruje rzeczone sygnały, może dojść do niekontrolowanego wzrostu komórek i rozwoju nowotworu[237].
Opisana współpraca w obrębie gatunku wyewoluowała dzięki doborowi krewniaczemu. Polega on na tym, że jeden organizm wspiera potomstwo swego krewnego[238]. Dobór naturalny premiuje takie zachowanie, jeśli pomagający osobnik posiada allele promujące niesienie pomocy i jest prawdopodobne, że jego krewniak również posiada te same allele, które zostaną przekazane dalej[239]. Inne procesy mogące promować współpracę to dobór grupowy, w przypadku którego współpraca dostarcza korzyści grupie organizmów[240].
Specjacja

Specjacja to proces, w którym gatunek rozdziela się na dwa bądź nawet więcej gatunków pochodnych[241].
Pojęcie gatunku zdefiniować można na wiele sposobów. Wybór definicji zależy od szczególnych cech rozważanych organizmów[242]. Na przykład pewne sformułowania pojęcia gatunku stosują się w bardziej czytelny sposób do organizmów rozmnażających się płciowo, podczas gdy inne lepiej opisują organizmy bezpłciowe. Pomimo zróżnicowania różnych koncepcji gatunku można je pogrupować w 3 kategorie, biorące pod uwagę rozród, ekologię i filogenetykę[243]. Biological Species Concept (BSC, koncept gatunku biologicznego) stanowi klasyczny przykład podejścia opartego na rozrodzie. Ernst Mayr w 1942 podał, że zgodnie z tym poglądem „gatunki są grupami rzeczywiście lub potencjalnie rozmnażających się ze sobą populacji naturalnych, izolowanych rozrodczo od innych takich grup”[244]. Pomimo szerokiego i długiego wykorzystywania BSC nie wyróżnia się od innych koncepcji brakiem kontrowersji. Na przykład nie można zastosować jej do prokariotów[245], co określa się problemem gatunku[242]. Niektórzy badacze podjęli starania unifikacji monistycznej definicji gatunku, podczas gdy inni adaptują podejście pluralistyczne i sugerują możliwość istnienia odmiennych sposobów logicznej interpretacji definicji gatunku[242][243].
Powstawanie nowego gatunku polega na powstawaniu barier dla przepływu genów (barier rozrodczych). Chodzi to u tworzenie się różnic biologicznych uniemożliwiających wspólny rozród (mechanizmów izolacji)[246]. Przepływ genów może spowolnić ten proces, rozprzestrzeniając nowe warianty genetyczne także w innych populacjach. Zależnie od tego, jak dalece dwa gatunki oddaliły się od swego ostatniego wspólnego przodka, mogą być jeszcze zdolne do wydawania mieszanego potomstwa, jak konie i osły, które mogą krzyżować się ze sobą. W efekcie na świat przychodzi muł[247]. Krzyżówki takie są zazwyczaj niepłodne. W tym przypadku blisko spokrewnione gatunki mogą regularnie parzyć się ze sobą, ale hybrydy będą eliminowane przez dobór naturalny i gatunki pozostaną odrębne. Jednak okazjonalnie pojawiają się żywotne krzyżówki i wtedy nowe gatunki miewają własności pośrednie pomiędzy gatunkami rodziców bądź też cechować się zupełnie odmiennym fenotypem[248]. Rola hybrydyzacji w tworzeniu się nowych gatunków zwierząt nie jest jasna, aczkolwiek przypadki takie obserwowano wśród wielu typów zwierząt[249]. Szczególnie dobrze zbadano to zjawisko na przykładzie rzekotki różnobarwnej[250].
Specjację obserwowano wielokrotnie w kontrolowanych warunkach laboratoryjnych oraz w naturze[251][252][253][254]. W przypadku organizmów rozmnażających się płciowo specjacja wynika z reprodukcyjnej izolacji, po której pojawia się zróżnicowanie genetyczne. Wyróżnia się mechanizmy specjacji. Najczęstszy to specjacja allopatryczna, zachodząca u gatunków izolowanych geograficznie, czy to na skutek fragmentacji siedlisk, czy to migracje. Dobór w tych warunkach może prowadzić do bardzo szybkich zmian w wyglądzie i zachowaniu organizmów[255][256]. Jako że dobór i dryf niezależnie działają na populację izolowaną od reszty gatunku, separacja może w końcu doprowadzić do powstania organizmów, które nie będą już w stanie krzyżować się z sobą[257].
Drugi mechanizm stanowi specjacja perypatryczna. Polega ona na izolacji niewielkiej grupy organizmów w nowym środowisku. Różnica dzieląca ją od specjacji allopatrycznej polega na znacznie mniejszej liczbie organizmów izolowanej populacji w porównaniu z populacją rodzicielską. Efekt założyciela powoduje szybką specjację po wzroście chowu wsobnego powodującym selekcję homozygot, co prowadzi do szybkich zmian genetycznych[258].
Trzeci mechanizm to specjacja parapatryczna. Przypomina on specjację perypatryczną w tym, że mała grupka osobników znajduje nowe siedlisko, jednakże w tym wypadku nie istnieje fizyczna bariera rozdzielająca organizmy tych dwu populacji. Zamiast tego specjacja pojawia się na skutek mechanizmów redukujących przepływ genów pomiędzy omawianymi populacjami[241]. Ogólnie zachodzi po drastycznej zmianie środowiskowej w siedlisku populacji rodzicielskiej. Przykładu dostarcza trawa tomka wonna, która mogła przejść specjację parapatryczną w odpowiedzi na zlokalizowane skażenie metalem[259]. Rośliny te wyewoluowały oporność na wysokie poziomy metali w glebie. Dobór przeciw krzyżowaniu się z wrażliwą na metal populacją rodzicielską doprowadził do stopniowych zmian w czasie kwitnienia opornych na metal roślin, co w końcu zaowocowało powstaniem całkowitej bariery rozrodczej. Selekcja przeciwko hybrydom dwu populacji może doprowadzić do wzmocnienia, ewolucji cech promujących rozród wewnątrz gatunku, jak rozsunięcie cech – oba gatunki stają się coraz bardziej różne od siebie nawzajem[260].
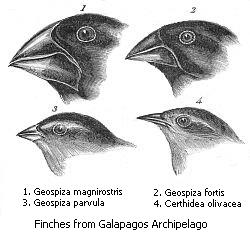
W końcu specjacja sympatryczna oznacza wyodrębnienie się gatunku bez izolacji geograficznej bądź zmiany w siedlisku. Ten rodzaj specjacji występuje rzadko, gdyż nawet niewielki przepływ genów może wyrównać różnice genetyczne pomiędzy częściami populacji[261][262]. Zazwyczaj specjacja sympatryczna u zwierząt wymaga ewolucji zarówno różnic genetycznych, jak i nielosowego dobierania się w pary, by mogła wyewoluować izolacja rozrodcza[263].
Jeden z rodzajów specjacji sympatrycznej wymaga krzyżowania się osobników dwóch blisko spokrewnionych ze sobą gatunków, by powstał kolejny gatunek. U zwierząt zjawisko to nie występuje często, a powstałe w taki sposób zwierzęce krzyżówki są zazwyczaj niepłodne. Powodem jest to, że w trakcie mejozy chromosomy homologiczne każdej pary pochodzą z dwóch różnych gatunków, wobec czego nie mogą się dokładnie sparować. U roślin zjawisko takie notuje się częściej, ponieważ rośliny często podwajają swój garnitur chromosomów, tworząc poliploidy[264]. Umożliwia to chromosomom każdego gatunku rodzicielskiego łączyć się ze sobą podczas mejozy, ponieważ od każdego z gatunków rodzicielskich pochodzi już para chromosomów[265]. Przykładu takiej specjacji dostarczają Arabidopsis thaliana i Arabidopsis arenosa których krzyżowanie dało początek nowemu gatunkowi Arabidopsis suecica[266]. Wydarzyło się to około 20 tys. lat temu[267], a proces specjacji udało się powtórzyć w laboratorium, co pozwoliło na zbadanie mechanizmów genetycznych zaangażowanych w ten proces[268]. W rzeczy samej podwajanie liczny chromosomów w obrębie gatunku może być często przyczyną izolacji reprodukcyjnej, gdyż połowa podwojonej liczby chromosomów nie będzie mogła połączyć się podczas rozrodu z połową wyjściowej ich liczby[269].
Procesy specjacji są ważne w teorii przerywanej równowagi, odpowiadającej wzorcowi zapisu kopalnego, przedstawiającego krótkotrwałe wybuchy ewolucji przerywane względnie długimi okresami stazy, podczas których szczątki względnie nie przedstawiają zmian[270]. Zgodnie z tą teorią specjacja wiąże się z szybką ewolucją, a dobór naturalny i dryf genetyczny działają najsilniej na organizmy przechodzące specjację w nowych siedliskach lub małych populacjach. W rezultacie okresy zastoju w zapisie kopalnym odpowiadają populacjom rodzicielskim, a organizmy przechodzące specjację i podlegające szybkiej ewolucji znajdują się w niewielkich populacjach na ograniczonych geograficznie siedliskach, wobec czego rzadko zachowują się jako skamieniałości[179].
Wymieranie

Wymieranie oznacza zniknięcie całego gatunku. Nie jest to wydarzenie niezwykłe, gatunki regularnie pojawiają się w procesie specjacji i zanikają, wymierając[271]. Prawie wszystkie gatunki roślin i zwierząt żyjące kiedykolwiek na Ziemi są już wymarłe[272], a wyginięcie wydaje się być ostatecznie losem każdego gatunku[273]. Wymierania zachodziły w sposób ciągły przez całą historię życia, choć tempo wymierania pikuje w epizodach masowego wymierania[274]. Najlepiej znane jest wymieranie kredowe, podczas którego wymarły między innymi nieptasie dinozaury. Jednak znacznie poważniejsze było wcześniejsze wymieranie permskie, kiedy to wymarło szacunkowo 96% wszystkich gatunków morskich[274]. Obecnie trwa wymieranie holoceńskie, związane z trwającą przez ostatnie kilka tysięcy lat ekspansją człowieka rozumnego. Obecne tempo wymierania przekracza 100–1000 razy bazowe tempo wymierania. Do połowy XXI wieku wyginąć może do 30% gatunków[275]. Jego główną przyczynę stanowi aktywność ludzka[276][277]; w przyszłości może przyśpieszyć je globalne ocieplenie[278].
Rola wymierania w ewolucji nie została jeszcze należycie zrozumiana. Może zależeć od rodzaju wymierania[274]. Przyczyny ciągłego wymierania, obejmującego większość wymierań gatunków, mogą wynikać z konkurencji między gatunkami i ograniczonych zasobów (zasada Gausego)[68]. Jeśli jeden gatunek może wygrać konkurencję z drugim, powstaje dobór, w wyniku którego lepiej dostosowany gatunek przeżywa, a drugi wymiera[137]. Przerywane masowe wymierania również są istotne. Nie działają jednak jako siła selekcyjna, ale drastycznie redukują różnorodność w sposób niespecyficzny gatunkowo, promując wybuchy szybkiej ewolucji i specjacji przetrwałych organizmów[279].
Ewolucyjna historia życia
Powstanie życia
Sama teoria ewolucji nie dotyczy powstania życia, zajmuje się tym teoria abiogenezy[280][281][282]. Z historycznej perspektywy teoria Darwina doprowadziła do rozwoju tej drugiej[283].
Ziemia liczy sobie około 4,54 miliarda lat[284][285][286]. Najwcześniejsze niedyskusyjne dowody istnienia życia na Ziemi datują się na co najmniej 3,5 miliarda lat[7][287], na czas eoarchaiku po rozpoczęciu się zestalania skorupy ziemskiej, ciekłej w poprzednim hadeiku. Skamieniałe pozostałości mat mikrobowych znaleziono w liczącym 3,48 miliarda lat piaskowcu w Australii Zachodniej[12][13][14]. Inne wczesne dowody fizyczne na obecność substancji biogennej obejmują grafit sprzed 3,7 miliarda lat z przekształconych metamorficznie skał osadowych Grenlandii Zachodniej[11], jak też pozostałości materiału biotycznego sprzed 4,1 miliarda lat z Australii Zachodniej[8][9].
Powyżej 99% wszystkich gatunków żyjących kiedykolwiek na Ziemi, których liczbę szacuje się na ponad 5 miliardów[288], wyginęło[289][17]. Szacunki liczby współczesnych gatunków podają od 10 do 15 milionów[18]. Dotychczas udokumentowano około 1,2 miliona, natomiast powyżej 86% z nich jeszcze nie odkryto[19].
Cząsteczki samoreplikujące się powstały dzięki chemii wysokoenergetycznej około 4 miliardów lat temu, pół miliarda lat przed ostatnim uniwersalnym wspólnym przodkiem[5]. Współczesny konsensus naukowy stanowi, że złożona biochemia, która dała początek życiu, powstała na skutek prostych reakcji chemicznych[290]. Powstanie życia obejmowało rozwinięcie się samoreplikujących molekuł, jak RNA[291], oraz prostych komórek[292].
Wspólne pochodzenie
Wszystkie zamieszkujące Ziemię organizmy pochodzą od jednego przodka czy wywodzą się z jednej puli genowej[206][293]. Współczesne gatunki cały czas podlegają ewolucji, a ich różnorodność wynika z długiej serii specjacji, którym towarzyszyły wymierania[294]. Istnienie wspólnego przodka organizmów wydedukowano wpierw z czterech prostych faktów. Po pierwsze rozmieszczenia geograficznego organizmów nie można wyjaśnić lokalną adaptacją. Po drugie różnorodności życia nie tworzy zbiór całkowicie unikatowych organizmów, jego elementy mają wspólne cechy (homologie). Po trzecie cechy szczątkowe bez jasnych funkcji przypominają funkcjonalne cechy organizmów ancestralnych. I w końcu organizmy można sklasyfikować w systemie hierarchicznym, przypominającym drzewo genealogiczne[295]. Jednak współczesne badania zasugerowały, że z powodu poziomego transferu genów drzewo życia może być bardziej skomplikowane, nie zawierać tylko pojedynczych odgałęzień; niektóre geny rozprzestrzeniły się niezależnie w odległych od siebie grupach organizmów[296][297].
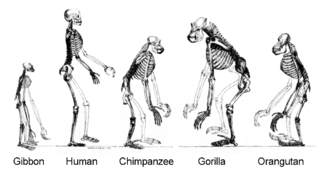
Minione gatunki pozostawiły zapis kopalny swej historii ewolucyjnej. Skamieliny, razem z anatomią porównawczą współczesnych organizmów, tworzą zapis morfologiczny czy też anatomiczny[298].
Bardziej współczesne dowody na wspólne pochodzenie biorą się z badań wykazujących biochemiczne podobieństwa pomiędzy organizmami. Przykładowo wszystkie żywe komórki bazują na tym samym zbiorze nukleotydów i aminokwasów[299]. Rozwój genetyki molekularnej ujawnił zapis ewolucji w genomie. Datowanie podziałów linii ewolucyjnych umożliwia zegar molekularny dzięki zachodzącym mutacjom[300]. Na przykład porównania sekwencji DNA ujawniło, że ludzie i szympansy dzielą 96% genomu. Analizy kilku obszarów różniących te rodzaje pomogło rzucić światło na czas życia ostatniego wspólnego przodka tych rodzajów[301], żyjącego około 4 milionów lat temu[302].
Ewolucja życia

Prokarioty pojawiły się na Ziemi około 3–4 miliardów lat temu[304][305][306]. Przez następne kilka miliardów lat nie zaszła żadna oczywista zmiana ich morfologii czy też organizacji ich komórki[307]. Komórka eukariotyczna powstała 1,6–2,7 miliarda lat temu. Ta duża zmiana struktury komórki zaszła, kiedy bakterie dostały się do wnętrza komórki i weszły z nią w związek zwany endosymbiozą[308][309]. Wewnątrzkomórkowe bakterie i ich gospodarz przeszły koewolucję, w wyniku której bakterie przekształciły się w mitochondria lub hydrogenosomy[310]. Z kolei dostanie się do wnętrza tych komórek organizmów przypominających sinice doprowadziło do powstania chloroplastów glonów i roślin[311][312].
Historia życia obejmowała jednokomórkowe eukarioty, prokarioty i archeony, zanim około 610 milionów lat temu (ediakar) w oceanach pojawiły się organizmy wielokomórkowe[304][313]. Ewolucja wielokomórkowości nastąpiła wielokrotnie u tak różnych grup organizmów, jak gąbki, brunatnice, sinice, bakterie śluzowe[314]. W styczniu 2016 doniesiono, że około 800 milionów lat temu niewielka zmiana genetyczna w pojedynczej cząsteczce GK-PID mogła umożliwić organizmom przejście od pojedynczej komórki do wielu komórek[315].
Niedługo po powstaniu pierwszych organizmów wielokomórkowych przez około 10 milionów lat znacząco wzrosła bioróżnorodność. Wydarzenie to określa się mianem eksplozji kambryjskiej. W zapisie kopalnym pojawiła się większość typów współczesnych zwierząt, a wraz z nimi wiele linii, które wyginęły[316]. Rozważanych jest wiele czynników, które mogły wywołać eksplozję kambryjską, w tym akumulacja tlenu w atmosferze na skutek fotosyntezy[317][318].
Około 500 milionów lat temu rośliny i grzyby skolonizowały lądy. Niedługo później ich śladem podążyły stawonogi i inne zwierzęta[319]. Owady odniosły wielki sukces ewolucyjny i do dziś stanowią większość gatunków zwierząt[320]. Płazy pojawiły się około 364 miliony lat temu, następnie zaś wczesne owodniowce. 155 milionów lat temu wyewoluowały ptaki. Ssaki powstały około 129 milionów lat temu. Homininae pojawiły się około 10 milionów lat temu, a człowiek anatomicznie współczesny – 0,25 miliona lat temu[321][322][323]. Pomimo ewolucji tych dużych zwierząt mniejsze organizmy żywe podobne do wczesnych form życia cały czas odnoszą sukces ewolucyjny i dominują na Ziemi, jako że większość biomasy tworzą prokarioty[183].
Zastosowania
Koncepcje i modele wykorzystywane w biologii ewolucyjnej, jak dobór naturalny, mają wiele zastosowań[324].
Dobór sztuczny to celowa selekcja cech w danej populacji organizmów. Przez tysiąclecia wykorzystywano go w procesie udomawiania roślin i zwierząt[325]. Bardziej współcześnie selekcja taka stała się ważną częścią inżynierii genetycznej. Geny selekcyjne (np. oporności na antybiotyki) służą w takich procesach do manipulacji DNA. Białka o cennych właściwościach wyewoluowały w powtarzanych cyklach mutacji i selekcji (na przykład zmodyfikowane enzymy i przeciwciała w procesie ewolucji kierowanej)[326].
Zrozumienie zmian, które zaszły w organizmach podczas ich ewolucji, może ujawnić geny konieczne dla budowy części ciała, które mogą być zaangażowane w choroby genetyczne[327]. Na przykład lustrzeń meksykański, albinotyczna ryba jaskiniowa, w procesie ewolucji utraciła oczy. Rozmnażanie ze sobą osobników z różnych populacji prowadzi czasami do powstania osobników z funkcjonalnymi oczami, jako że w izolowanych populacjach różnych jaskiń zaszły różne mutacje[328]. Pomogło to zidentyfikować geny konieczne dla widzenia i pigmentacji[329].
Wiele chorób człowieka to nie stałe zjawisko, lecz podlegają one ewolucji. Wirusy, bakterie, grzyby i nowotwory ewoluują, wytwarzając oporność przeciw układowi odpornościowemu i lekom[330][331][332]. Te same problemy pojawiają się w rolnictwie w przypadku pestycydów[333] i herbicydów[334]. Możliwe, że efektywne użytkowanie antybiotyków dobiega końca[335]. Przewiduje się, że ewolucja i ewoluowalność[336] patogenów wiążą się z koniecznością opracowywania strategii spowolnienia bądź przechytrzenia tego procesu, wymaga to jednak głębszej wiedzy o złożonych procesach rządzących ewolucją na poziomie cząsteczkowym[337].
W informatyce symulacje ewolucji z wykorzystaniem algorytmów ewolucyjnych i sztucznego życia rozpoczęły się w latach sześćdziesiątych XX wieku, rozwijano je, symulując dobór sztuczny[338]. Sztuczna ewolucja stała się szeroko rozpowszechnionym sposobem optymizacji w efekcie prac Ingo Rechenberga w siódmej dekadzie XX wieku. Wykorzystał on strategie ewolucyjne do rozwiązywania złożonych problemów inżynieryjnych[339]. Algorytmy genetyczne stały się popularne w szczególności dzięki dziełom Hollanda[340]. Praktyczne aplikacje obejmują również automatyczną ewolucję programów komputerowych[341]. Algorytmy ewolucyjne obecnie wykorzystywane są do rozwiązywania wielowymiarowych problemów wydajniej, niż oprogramowanie stworzone przez człowieka i optymalizują tworzenie systemów[342].
Percepcja i krytyka teorii ewolucji

Stopniowe przyjęcie ewolucji przez środowiska uczonych z nurtu chrześcijańskiego
W XIX wieku, szczególnie po publikacji On the Origin of Species w 1859, temat ewolucji stanowił najgłośniejsze zagadnienie w środowiskach naukowych Europy[344]. Nie bez znaczenia były także filozoficzne, społeczne i teologiczne implikacje teorii Darwina. Jednym z ich krytyków był brytyjski biolog St. George Mivart, który napisał książkę On the Genesis of Species (O pochodzeniu gatunków). Z powodów religijnych, ale także rozumowych, odrzucił on m.in. pogląd o naturalnej selekcji. Pisał o tym, podsumowując wnioski płynące ze swej refleksji opisanej w książce:
...głębsze przemyślenie i bardziej uważne badanie ukazywało Autorowi teorię pana Darwina jako coraz bardziej niewystarczającą, jeśli chodzi o zachowanie i wzmocnienie początkowego, gatunkowego i rodzajowego charakteru cech. Twierdzenie, że te drobne, korzystne i nieokreślone różnice mogłyby wywołać tak szczególne formy i modyfikacje (...) zdaje się sprzeciwiać nie wyobraźni, lecz rozumowi[345].
Już reakcje na publikację Vestiges of the Natural History of Creation w 1844 r. pokazały, że największe kontrowersje w biologii ewolucyjnej budzi ewolucja człowieka – to, że człowiek dzieli wspólne pochodzenie z małpami człekokształtnymi. Możliwe konsekwencje dla ludzkiej moralności byłyby takie, że posiada ona takie same naturalne przyczyny, jak inne dziedziczne cechy występujące u wszystkich zwierząt[346]. Rzeczywiście, jak zauważył Giorgio Manzi, mimo iż w The Origin of Species (1859 r.) Darwina, na temat człowieka znajduje się jedynie jedno zdanie pod koniec książki: „światło będzie rzucone na początek człowieka i jego historię”[347], ożywiona dyskusja zaczęła się toczyć nie na temat orchidei, zięb czy żółwi, lecz skupiła się na pokrewieństwie małp i człowiekowatych do Homo sapiens[348].
Współczesna synteza ewolucyjna doprowadziła do ujednolicenia biologii w okresie 1930–1950, co wiązało się z szerokim konsensusem w zakresie mechanizmów wywołujących zmiany ewolucyjne. Poszerzanie i rozwój wiedzy doprowadził takich autorów jak G.L. Stebbins, Th. Dobzhansky (genetycy), E. Mayr, J. Huxley (zoologowie), G. Gaylord Simpson, G.L. Jepsen (paleontolodzy), R.A. Fisher i S. Wright (genetycy matematyczni) do sformułowania w 1941 r. nowej wersji darwinizmu, w postaci współczesnej syntezy ewolucyjnej. Miała ona taką siłę, że stała się odtąd unikatową, jednoczącą zasadą biologii[349]. Uczeni ci, oprócz tematów czysto biologicznych, w swych wypowiedziach na temat ewolucji zwracają się przeciw tradycyjnemu w kulturze europejskiej – ukształtowanej przez judeo-chrześcijaństwo – poglądowi o stworzeniu świata przez Boga. Uznają jednoznacznie, że powstanie życia jest skutkiem ślepych, przypadkowych procesów.[potrzebny przypis] Julian Huxley, odwracając obraz Boga, który stwarza człowieka mówił m.in. o Bogu stworzonym przez wyobraźnię człowieka. Również religię postrzegał jako produkt człowieka, mający zaspokoić odpowiednie jego potrzeby. Przewidywał też wytworzenie nowej religii, dostosowanej do mentalności ewolucjonistycznej:
Człowiek ewolucyjny nie może już nadal uciekać od swej samotności w ramiona istniejącego tylko w wyobraźni boskiego ojca, którego sam stworzył (...). Wizja ewolucyjna umożliwia nam dostrzeżenie, przynajmniej w zarysie, cechy nowej religii, jaka z pewnością powstanie, by zaspokajać potrzeby nadchodzącej epoki[350][351].
Najgłośniejszym współczesnym teoretykiem tego nurtu, świadomie podkreślającym ateistyczną interpretację teorii ewolucji, jest Richard Dawkins, który napisał m.in. Ślepy zegarmistrz, czyli jak ewolucja dowodzi, że świat nie został zaplanowany[352] oraz Bóg urojony[353].
W dokumencie „Kościół wobec ewolucji” wydanym przez Radę Naukową Episkopatu Polski w listopadzie 2006, biskupi przywołują słowa Jana Pawła II z 1996 roku: „Nowe zdobycze nauki każą nam uznać, że teoria ewolucji jest czymś więcej niż hipotezą”. Krytykują też przedstawicieli „fideistycznego kreacjonizmu, którzy interpretują dosłownie biblijne opowiadanie o stworzeniu świata [...] i na tej podstawie kwestionują nauczanie o ewolucji w szkole”. Uznają też, że „fundamentalistyczny kreacjonizm nie jest zgodny z nauką katolicką”[354].
Różne wyznania dokonały prób integracji ewolucji ze swoją doktryną religijną. Uznaje się, że niekoniecznie ewolucja musi wychodzić z przesłanek ateistycznych. Tak ujmuje ją tzw. teistyczny ewolucjonizm, który także wśród teologów katolickich znalazł umiarkowane poparcie[355].
Opozycja kreacjonistów wobec teorii ewolucji
Tezy biologii ewolucyjnej są jednak nadal kwestionowane przez zwolenników inteligentnego projektu i innych wersji kreacjonizmu[68]. Synteza ewolucyjna pozostaje sporna dla niektórych teistów[356][357][346][358].
Istnieją dwa nurty kreacjonistów, które sprzeciwiają się teorii ewolucji. Pierwszy nurt to tzw. konkordyzm kreacjonistyczny, próbujący sformułować teorię powstania Ziemi, w całkowitej zgodności z literalnym odczytaniem pierwszych rozdziałów Księgi Rodzaju. Drugi nurt to tzw. kreacjonizm naukowy, rozwijający się w środowisku ortodoksyjnych protestantów drugiej połowy XX w. Wychodzili oni z przekonania, że Bóg objawił się w swoim Słowie, które należy przyjąć takim, jakie ono jest. Niewykluczone, że nauka kiedyś to Słowo potwierdzi. Z tego nurtu wyrósł następnie ruch inteligentnego projektu[359].
W niektórych krajach, zwłaszcza w USA, napięcia pomiędzy nauką a religią doprowadziły do konfliktu skupiającego się na polityce i edukacji publicznej[360]. Chociaż inne dziedziny nauki, jak kosmologia[361] czy nauki o Ziemi[362], również stoją w konflikcie z literalnym odczytem wielu tekstów religijnych, to antyewolucjonizm, z kreacjonizmem na czele, jest silny i dobrze zorganizowany[363].
Spór między kreacjonistami a ewolucjonistami rozpoczął się w latach dwudziestych XX wieku jako efekt uboczny konfliktu w ramach amerykańskiego protestantyzmu. Obóz określany jako fundamentaliści ukształtował się w efekcie historycznej krytyki Biblii, postępu technicznego i społecznego oraz wiedzy o ewolucji. Drugi obóz – moderniści – traktowali Biblię jako tekst alegoryczny. Konflikt wybuchł w pełni po I wojnie światowej, gdy polityk William Jennings Bryan uznał, że „darwinizm” był teoretyczną podstawą niemieckiego militaryzmu i jest zagrożeniem dla tradycyjnej religii i moralności. Efektem kampanii społecznej był zakaz nauczania o ewolucji w kilku stanach, a zagadnienia ewolucyjne szybko zniknęły z podręczników szkolnych[175]. Nauczanie o ewolucji w amerykańskiej szkole pozostało rzadkością przez większą część pierwszej połowy XX wieku. Decyzja Scopes Trial z 1925 spowodowała, że wiedza o ewolucji stała się jeszcze większą rzadkością w podręcznikach biologii przez całe pokolenie, ale później była stopniowo przywracana[364]. W latach pięćdziesiątych XX wieku strach przed technologiczną przewagą Związku Radzieckiego doprowadził do sfinansowania z budżetu federalnego nowych podręczników napisanych przez naukowców. Zawierały one ponownie wiedzę o ewolucji biologicznej[175]. W 1968 biologia ewolucyjna została objęta ochroną prawną przez postanowienie w sprawie Epperson v. Arkansas. Odtąd konkurujące z ewolucjonizmem wierzenia religijne, a więc kreacjonizm, zostały prawnie wycofane ze szkół drogą różnych postanowień w latach siedemdziesiątych i osiemdziesiątych XX wieku. Powróciły jednak w pseudonaukowej formie inteligentnego projektu, by zostać wycofane raz jeszcze w 2005 po sprawie Kitzmiller v. Dover Area School District[364].
Kwestia przypadkowości ewolucji
Ewolucja wcale nie musi zwiększać złożoności[365][366]. Celowościowa, uporządkowana wizja przyrody przemawiała i przemawia zarówno do filozofów, jak i przyrodników. Pod koniec XIX wieku Hugh Miller proponował jako cel ewolucji centralną pozycję ssaków wśród istot żywych[367]. Teorie ortogenezy, jednej linii rozwojowej, stanowiły, że dany gatunek ewoluuje w określonym kierunku. Negowały one rolę doboru w ewolucji, nie proponowały jednak w zamian żadnego mechanizmu mającego wyjaśniać postulowane procesy[368]. Według opinii Christopha Schönborna, wyrażonej w artykule zamieszczonym w dzienniku New York Times w 2005 r., ślepa selekcja nie jest wystarczającym wyjaśnieniem skomplikowanych procesów, które nauka dostrzega w organizmach żywych i trzymanie się tej teorii przeczy faktom i wypływa z postaw ideowych, a nie naukowych:
Ewolucja w sensie neodarwinowskim – jako niekierowany, nieplanowany proces przypadkowych zmian i naturalnej selekcji – nie jest /prawdziwa/. Każdy system myśli, który zaprzecza lub dąży do opacznego wyjaśnienia przytłaczających dowodów na rzecz istnienia projektu w biologii jest ideologią, a nie nauką[369].
Według wiedeńskiego teologa świat nie przedstawia chaosu lub przypadku; celowość i plan są zbyt dobrze widoczne i potwierdzone przez naukę, na co mogą się w jego opinii zgodzić nie tylko wierzący, ale i ateiści[369][370]. W odpowiedzi na dyskusję, którą wywołał jego głos w nowojorskiej gazecie, Schönborn wygłosił w katedrze wiedeńskiej cykl katechez, w których wyjaśnił swoje przekonanie o możliwości odnalezienia racjonalnego porządku w historii świata i życia. Zostały opublikowane w książce, wydanej także w języku polskim, pt. Cel czy przypadek? Dzieło stworzenia i ewolucja z punktu widzenia racjonalnej wiary[371]. Jego poglądy stoją w sprzeczności z poglądami niektórych innych myślicieli. Na przykład Michał Heller zwraca uwagę na ważne miejsce przypadku w działaniu świata, stanowiącego jego integralną część, podlegającego działaniu praw rządzących tym światem i opisywalnego matematycznie[372].
Zasady termodynamiki a ewolucja
Argumentem stosowanym przeciw ewolucji, popularyzowanym przez Kenta Hovinda[373], jest stwierdzenie jakoby druga zasada termodynamiki przeczyła ewolucji[374]. W rzeczywistości żadna sprzeczność nie występuje, ponieważ Ziemia dostaje energię ze Słońca. Gdyby traktować Ziemię jako układ izolowany, kryształki minerałów i płatki śniegu również byłyby niemożliwe, ponieważ one również są złożonymi strukturami, które tworzą się samoistnie z nieuporządkowanych części[375].
Przypisy
- ↑ Futuyma 2008 ↓, s. 2.
- ↑ Hall i Hallgrímsson 2008 ↓, s. 3–5.
- ↑ a b c R.C. Lewontin. The Units of Selection. „Annual Review of Ecology and Systematics”. 1, s. 1–18, 1970. Palo Alto, CA: Annual Reviews. DOI: 10.1146/annurev.es.01.110170.000245. ISSN 1545-2069. JSTOR: 2096764.
- ↑ Kampourakis 2014 ↓, s. 127–129.
- ↑ a b W. Ford Doolittle. Uprooting the Tree of Life. „Scientific American”. 282 (2), s. 90–95, 2000. Stuttgart: Georg von Holtzbrinck Publishing Group. DOI: 10.1038/scientificamerican0200-90. ISSN 0036-8733. PMID: 10710791. [dostęp 2015-04-05]. [zarchiwizowane z adresu 2006-09-07].
- ↑ Nicolas Glansdorff, Ying Xu, Bernard Labedan. The Last Universal Common Ancestor: emergence, constitution and genetic legacy of an elusive forerunner. „Biology Direct”. 3, s. 29, 2008-07-09. London: BioMed Central. DOI: 10.1186/1745-6150-3-29. ISSN 1745-6150. PMID: 18613974. PMCID: PMC2478661.
- ↑ a b J. William Schopf, Anatoliy B. Kudryavtsev, Andrew D. Czaja, Abhishek B. Tripathi. Evidence of Archean life: Stromatolites and microfossils. „Precambrian Research”. 158 (3–4), s. 141–155, 2007-10-05. Amsterdam, Holandia: Elsevier. DOI: 10.1016/j.precamres.2007.04.009. ISSN 0301-9268.
- ↑ a b Seth Borenstein. Hints of life on what was thought to be desolate early Earth. „Excite”, 2015. Yonkers, NY: Mindspark Interactive Network. [dostęp 2015-10-20].
- ↑ a b Elizabeth A. Bell, Patrick Boehnike, T. Mark Harrison, Wendy L. Mao. Potentially biogenic carbon preserved in a 4.1 billion-year-old zircon. „Proc. Natl. Acad. Sci. U.S.A.”. 112 (47), s. 14518–14521, 2015-11-24. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.1517557112. ISSN 0027-8424. PMID: 26483481. PMCID: PMC4664351. [dostęp 2015-12-30].
- ↑ Panno 2005 ↓, s. xv-16.
- ↑ a b Yoko Ohtomo, Takeshi Kakegawa, Akizumi Ishida, Toshiro Nagase i inni. Evidence for biogenic graphite in early Archaean Isua metasedimentary rocks. „Nature Geoscience”. 7 (1), s. 25–28, 2014. London: Nature Publishing Group. DOI: 10.1038/ngeo2025. ISSN 1752-0894. Bibcode: 2014NatGe...7...25O.
- ↑ a b Seth Borenstein. Oldest fossil found: Meet your microbial mom. „Excite”, 2013-11-13. Yonkers, NY: Mindspark Interactive Network. [dostęp 2015-05-31].
- ↑ a b Jonathan Pearlman. 'Oldest signs of life on Earth found’. „The Daily Telegraph”, 13 listopada 2013. London: Telegraph Media Group. [dostęp 2014-12-15].
- ↑ a b Nora Noffke, Daniel Christian, David Wacey, Robert M. Hazen. Microbially Induced Sedimentary Structures Recording an Ancient Ecosystem in the ca. 3.48 Billion-Year-Old Dresser Formation, Pilbara, Western Australia. „Astrobiology”. 13 (12), s. 1103–1124, 2013-11-16. New Rochelle, NY: Mary Ann Liebert, Inc.. DOI: 10.1089/ast.2013.1030. ISSN 1531-1074. PMID: 24205812. PMCID: PMC3870916. Bibcode: 2013AsBio..13.1103N.
- ↑ Futuyma 2004 ↓, s. 33.
- ↑ Stearns i Stearns 1999 ↓, s. x.
- ↑ a b Michael J. Novacek. Prehistory’s Brilliant Future. „The New York Times”, 2014-11-08. New York: The New York Times Company. ISSN 0362-4331. [dostęp 2014-12-25].
- ↑ a b Miller i Spoolman 2012 ↓, s. 62.
- ↑ a b Camilo Mora, Derek P. Tittensor, Sina Adl, Alastair G.B. Simpson i inni. How Many Species Are There on Earth and in the Ocean?. „PLOS Biology”. 9 (8), s. e1001127, 23 sierpnia 2011. San Francisco, CA: Public Library of Science. DOI: 10.1371/journal.pbio.1001127. ISSN 1545-7885. PMID: 21886479. PMCID: PMC3160336.
- ↑ Darwin 1859 ↓, s. Chapter XIV.
- ↑ Thomas C. Scott-Phillips, Kevin N. Laland, D.M. Shuker, Thomas E. Dickins i inni. The Niche Construction Perspective: A Critical Appraisal. „Evolution”. 68 (5). s. 1231–1243. DOI: 10.1111/evo.12332. ISSN 1558-5646.
- ↑ Provine 1988 ↓, s. 49–79.
- ↑ National Academy of Sciences, Institute of Medicine: Science, Evolution, and Creationism. Washington, D.C.: National Academy Press, 2008, s. R11–R12. ISBN 978-0-309-10586-6. LCCN 2007015904. OCLC 123539346. (ang.).
- ↑ Ayala i Avise 2014 ↓.
- ↑ National Academy of Sciences, Institute of Medicine: Science, Evolution, and Creationism. Washington, D.C.: National Academy Press, 2008, s. 17. ISBN 978-0-309-10586-6. LCCN 2007015904. OCLC 123539346. (ang.).
- ↑ Moore, Decker i Cotner 2010 ↓, s. 454.
- ↑ Douglas J. Futuyma, Evolution, Science, and Society: Evolutionary Biology and the National Research Agenda, New Brunswick, NJ: Office of University Publications, Rutgers, The State University of New Jersey, 1999, OCLC 43422991 [dostęp 2016-12-09] [zarchiwizowane z adresu 2012-01-31].
- ↑ Kirk, Raven i Schofield 1983 ↓, s. 100–142, 280–321.
- ↑ Book V, lines 855–877. W: Lucretius: De Rerum Natura. Edited and translated by William Ellery Leonard (1916). Medford/Somerville, MA: Tufts University. OCLC 33233743. [dostęp 2014-11-25].
- ↑ David Sedley. Lucretius and the New Empedocles. „Leeds International Classical Studies”. 2 (4), 2003. Leeds, West Yorkshire, England: Leeds International Classics Seminar. ISSN 1477-3643. [dostęp 2014-11-25]. [zarchiwizowane z adresu].
- ↑ Harry Beal Torrey, Frances Felin. Was Aristotle an Evolutionist?. „The Quarterly Review of Biology”. 12 (1), s. 1–18, 1937. DOI: 10.1086/394520. ISSN 0033-5770. JSTOR: 2808399.
- ↑ David L. Hull. The Metaphysics of Evolution. „The British Journal for the History of Science”. 3 (4), s. 309–337, grudzień 1967. Cambridge: Cambridge University Press. DOI: 10.1017/S0007087400002892. JSTOR: 4024958.
- ↑ Mason 1962 ↓, s. 43–44.
- ↑ Michał Heller, Tadeusz Pabjan, Stworzenie i początek wszechświata, Kraków: Copernicus Center Press, 2016, s. 64, Cytat: Wielu autorów dopatruje się w jego [św. Augustyna] koncepcji zapowiedzi albo nawet swoiście pojmowanego pierwowzoru teorii ewolucji.
- ↑ Michał Heller, Tadeusz Pabjan, Stworzenie i początek wszechświata, Kraków: Copernicus Center, 2016, s. 63–64.
- ↑ Mayr 1982 ↓, s. 256–257.
- ↑ Ray 1686 ↓.
- ↑ Bowler 2003 ↓, s. 73–75.
- ↑ Erasmus Darwin (1731–1802). „Evolution”, 1995-10-04. Berkeley, CA: University of California Museum of Paleontology. [dostęp 0201-02-112].
- ↑ Lamarck 1809 ↓.
- ↑ a b Nardon i Grenier 1991 ↓, s. 162.
- ↑ a b c Gould 2002 ↓.
- ↑ Michael T. Ghiselin. The Imaginary Lamarck: A Look at Bogus ‘History’ in Schoolbooks. „The Textbook Letter”, wrzesień–październik 1994. Sausalito, CA: The Textbook League. OCLC 23228649. [dostęp 2008-01-23]. [zarchiwizowane z adresu].
- ↑ Magner 2002 ↓.
- ↑ Eva Jablonka, Marion J. Lamb. Précis of Evolution in Four Dimensions. „Behavioural and Brain Sciences”. 30 (4), s. 353–365, 2007. Cambridge: Cambridge University Press. DOI: 10.1017/S0140525X07002221. ISSN 0140-525X.
- ↑ Burkhardt i Smith 1991 ↓.
- ↑ Darwin, C. R. to Lubbock, John, Darwin Correspondence Project, Cambridge, UK: University of Cambridge [dostęp 2014-12-01]. Letter 2532, November 22, 1859.
- ↑ Frank J. Sulloway. Why Darwin rejected intelligent design. „Journal of Biosciences”. 34 (2), s. 173–183, 2009. Bangalore: Indian Academy of Sciences. DOI: 10.1007/s12038-009-0020-8. ISSN 0250-5991. PMID: 19550032.
- ↑ Dawkins, 1990.
- ↑ Elliott Sober. Did Darwin write the Origin backwards?. „Proc. Natl. Acad. Sci. U.S.A.”. 106 (Suppl. 1), s. 10048–10055, 16 czerwca 2009. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0901109106. ISSN 0027-8424. Bibcode: 2009PNAS..10610048S.
- ↑ Mayr 2002 ↓, s. 165.
- ↑ Bowler 2003 ↓, s. 145–146.
- ↑ Robert R. Sokal, Theodore J. Crovello. The Biological Species Concept: A Critical Evaluation. „The American Naturalist”. 104 (936), s. 127–153, 1970. Chicago, IL: University of Chicago Press on behalf of the American Society of Naturalists. DOI: 10.1086/282646. ISSN 0003-0147. JSTOR: 2459191.
- ↑ Józef Życiński: U źródeł biologii niearystotelesowskiej. W: Michał Heller, Józef Życiński: Dylematy ewolucji. Kraków: Copernicus Press Center, 2016, s. 52, seria: Nauka i Religia. ISBN 978-83-7886-212-3.
- ↑ Charles Darwin, Alfred Wallace. On the Tendency of Species to form Varieties; and on the Perpetuation of Varieties and Species by Natural Means of Selection. „Journal of the Proceedings of the Linnean Society of London. Zoology”. 3 (9), s. 45–62, 1858-08-20. DOI: 10.1111/j.1096-3642.1858.tb02500.x. ISSN 1096-3642. [dostęp 2007-05-13].
- ↑ Thomas Henry Huxley, [w:] Encyclopædia Britannica [dostęp 2014-12-02] (ang.).
- ↑ Y.S. Liu, X.M. Zhou, M.X. Zhi, X.J. Li i inni. Darwin’s contributions to genetics. „Journal of Applied Genetics”. 50 (3), s. 177–184, 2009. Poznań: Instytut Genetyki Roślin, Polska Akademia Nauk. DOI: 10.1007/BF03195671. ISSN 1234-1983. PMID: 19638672.
- ↑ Franz Weiling. Historical study: Johann Gregor Mendel 1822–1884. „American Journal of Medical Genetics”. 40 (1), s. 1–26, lipiec 1991. DOI: 10.1002/ajmg.1320400103. PMID: 1887835.
- ↑ Wright 1984 ↓, s. 480.
- ↑ Provine 1971 ↓.
- ↑ Ida H. Stamhuis, Onno G. Meijer, Erik J.A. Zevenhuizen. Hugo de Vries on Heredity, 1889–1903: Statistics, Mendelian Laws, Pangenes, Mutations. „Isis”. 90 (2), s. 238–267, 1999. Chicago, IL: University of Chicago Press. DOI: 10.1086/384323. ISSN 0021-1753. PMID: 10439561. JSTOR: 237050.
- ↑ Quammen 2006 ↓.
- ↑ Bowler 1989 ↓.
- ↑ J.D. Watson, F.H.C. Crick. Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid. „Nature”. 171 (4356), s. 737–738, 1953-04-25. London: Nature Publishing Group. DOI: 10.1038/171737a0. ISSN 0028-0836. PMID: 13054692. Bibcode: 1953Natur.171..737W. [dostęp 2014-12-04]. Cytat: It has not escaped our notice that the specific pairing we have postulated immediately suggests a possible copying mechanism for the genetic material..
- ↑ Hennig 1999 ↓, s. 280.
- ↑ Wiley i Lieberman 2011 ↓.
- ↑ Theodosius Dobzhansky. Nothing in Biology Makes Sense Except in the Light of Evolution. „The American Biology Teacher”. 35 (3), s. 125–129, 1973. McLean, VA: National Association of Biology Teachers. DOI: 10.2307/4444260.
- ↑ a b c Ulrich Kutschera, Karl J. Niklas. The modern theory of biological evolution: an expanded synthesis. „Naturwissenschaften”. 91 (6), s. 255–276, czerwiec 2004. Heidelberg: Springer-Verlag Heidelberg. DOI: 10.1007/s00114-004-0515-y. ISSN 1432-1904. PMID: 15241603. Bibcode: 2004NW.....91..255K.
- ↑ Cracraft i Bybee 2005 ↓.
- ↑ John C. Avise, Francisco J. Ayala. In the light of evolution IV: The human condition. „Proc. Natl. Acad. Sci. U.S.A.”. 107 (Suppl. 2), s. 8897–8901, 11 maja 2010. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.100321410. ISSN 0027-8424. [dostęp 2014-12-29].
- ↑ A Commentary on Blute’s ‘Updated Definition’, Denis Walsh, 2009.
- ↑ Richard A. Sturm, Tony N. Frudakis. Eye colour: portals into pigmentation genes and ancestry. „Trends in Genetics”. 20 (8), s. 327–332, sierpień 2004. Cambridge, MA: Cell Press. DOI: 10.1016/j.tig.2004.06.010. ISSN 0168-9525. PMID: 15262401.
- ↑ a b Helen Pearson. Genetics: What is a gene?. „Nature”. 441 (7092), s. 398–401, 2006-05-25. London: Nature Publishing Group. DOI: 10.1038/441398a. ISSN 0028-0836. PMID: 16724031. Bibcode: 2006Natur.441..398P.
- ↑ a b Futuyma 2008 ↓, s. 10.
- ↑ Peter M. Visscher, William G. Hill, Naomi R. Wray. Heritability in the genomics era – concepts and misconceptions. „Nature Reviews Genetics”. 9 (4), s. 255–266, 2008. London: Nature Publishing Group. DOI: 10.1038/nrg2322. ISSN 1471-0056. PMID: 18319743.
- ↑ William S. Oetting, Murray H. Brilliant, Richard A. King. The clinical spectrum of albinism in humans. „Molecular Medicine Today”. 2 (8), s. 330–335, 1996. Cambridge, MA: Cell Press. DOI: 10.1016/1357-4310(96)81798-9. ISSN 1357-4310. PMID: 8796918.
- ↑ a b Futuyma, 2005.
- ↑ Patrick C. Phillips. Epistasis–the essential role of gene interactions in the structure and evolution of genetic systems. „Nature Reviews Genetics”. 9 (11), s. 855–867, listopad 2008. London: Nature Publishing Group. DOI: 10.1038/nrg2452. ISSN 1471-0056. PMID: 18852697. PMCID: PMC2689140.
- ↑ a b Rongling Wu, Min Lin. Functional mapping – how to map and study the genetic architecture of dynamic complex traits. „Nature Reviews Genetics”. 7 (3), s. 229–237, 2006. London: Nature Publishing Group. DOI: 10.1038/nrg1804. ISSN 1471-0056. PMID: 16485021.
- ↑ Eva Jablonka, Gal Raz. Transgenerational Epigenetic Inheritance: Prevalence, Mechanisms, and Implications for the Study of Heredity and Evolution. „The Quarterly Review of Biology”. 84 (2), s. 131–176, 2009. Chicago, IL: University of Chicago Press. DOI: 10.1086/598822. ISSN 0033-5770. PMID: 19606595.
- ↑ Oliver Bossdorf, Davide Arcuri, Christina L. Richards, Massimo Pigliucci. Experimental alteration of DNA methylation affects the phenotypic plasticity of ecologically relevant traits in Arabidopsis thaliana. „Evolutionary Ecology”. 24 (3), s. 541–553, 2010. Dordrecht, the Netherlands: Springer Netherlands. DOI: 10.1007/s10682-010-9372-7. ISSN 0269-7653.
- ↑ Jablonka i Lamb 2005 ↓.
- ↑ Eva Jablonka, Marion J. Lamb. The Changing Concept of Epigenetics. „Annals of the New York Academy of Sciences”. 981 (1), s. 82–96, 2002. Hoboken, NJ: Wiley-Blackwell on behalf of the New York Academy of Sciences. DOI: 10.1111/j.1749-6632.2002.tb04913.x. ISSN 0077-8923. PMID: 12547675. Bibcode: 2002NYASA.981...82J.
- ↑ Kevin N. Laland, Kim Sterelny. Perspective: Seven Reasons (Not) to Neglect Niche Construction. „Evolution”. 60 (9), s. 1751–1762, wrzesień 2006. Hoboken, NJ: John Wiley & Sons, Society for the Study of Evolution. DOI: 10.1111/j.0014-3820.2006.tb00520.x. ISSN 0014-3820.
- ↑ Michael J. Chapman, Lynn Margulis. Morphogenesis by symbiogenesis. „International Microbiology”. 1 (4), s. 319–326, grudzień 1998. Barcelona: Spanish Society for Microbiology. ISSN 1139-6709. PMID: 10943381. [dostęp 2014-12-09].
- ↑ David Sloan Wilson, Edward O. Wilson. Rethinking the Theoretical Foundation of Sociobiology. „The Quarterly Review of Biology”. 82 (4), s. 327–348, 2007. Chicago, IL: University of Chicago Press. DOI: 10.1086/522809. ISSN 0033-5770. PMID: 18217526. [zarchiwizowane z adresu].
- ↑ William Amos, John Harwood. Factors affecting levels of genetic diversity in natural populations. „Philosophical Transactions of the Royal Society B: Biological Sciences”. 353 (1366), s. 177–186, 1998-02-28. London: Royal Society. DOI: 10.1098/rstb.1998.0200. ISSN 0962-8436. PMID: 9533122. PMCID: PMC1692205.
- ↑ a b W. J. Ewens, 2004.
- ↑ Roger K. Butlin, Tom Tregenza. Levels of genetic polymorphism: marker loci versus quantitative traits. „Philosophical Transactions of the Royal Society B: Biological Sciences”. 353 (1366), s. 187–198, 28 lutego 1998. London: Royal Society. DOI: 10.1098/rstb.1998.0201. ISSN 0962-8436. PMID: 9533123. PMCID: PMC1692210.
- ↑ Roger K. Butlin, Tom Tregenza. Correction for Butlin and Tregenza, Levels of genetic polymorphism: marker loci versus quantitative traits. „Philosophical Transactions of the Royal Society B: Biological Sciences”. 355 (1404), s. 1865, 29 grudnia 2000. London: Royal Society. DOI: 10.1098/rstb.2000.2000. ISSN 0962-8436. Cytat: Some of the values in table 1 on p. 193 were given incorrectly. The errors do not affect the conclusions drawn in the paper. The corrected table is reproduced below..
- ↑ Anna Wetterbom, Marie Sevov, Lucia Cavelier, Tomas F. Bergström. Comparative Genomic Analysis of Human and Chimpanzee Indicates a Key Role for Indels in Primate Evolution. „Journal of Molecular Evolution”. 63 (5), s. 682–690, 2006. New York: Springer-Verlag New York. DOI: 10.1007/s00239-006-0045-7. ISSN 0022-2844. PMID: 17075697.
- ↑ Stanley A. Sawyer, John Parsch, Zhang Zhi, Daniel L. Hartl. Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila. „Proc. Natl. Acad. Sci. U.S.A.”. 104 (16), s. 6504–6510, 2007-04-17. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0701572104. ISSN 0027-8424. PMID: 17409186. PMCID: PMC1871816. Bibcode: 2007PNAS..104.6504S.
- ↑ P.J. Hastings, James R. Lupski, Susan M. Rosenberg, Grzegorz Ira. Mechanisms of change in gene copy number. „Nature Reviews Genetics”. 10 (8), s. 551–564, 2009. London: Nature Publishing Group. DOI: 10.1038/nrg2593. ISSN 1471-0056. PMID: 19597530. PMCID: PMC2864001.
- ↑ Carroll, Grenier i Weatherbee 2005 ↓.
- ↑ Paul M. Harrison, Mark Gerstein. Studying Genomes Through the Aeons: Protein Families, Pseudogenes and Proteome Evolution. „Journal of Molecular Biology”. 318 (5), s. 1155–1174, 17 maja 2002. Amsterdam, the Netherlands: Elsevier. DOI: 10.1016/S0022-2836(02)00109-2. ISSN 0022-2836. PMID: 12083509.
- ↑ James K. Bowmaker. Evolution of colour vision in vertebrates. „Eye”. 12 (3b), s. 541–547, 1998. London: Nature Publishing Group on behalf of the Royal College of Ophthalmologists. DOI: 10.1038/eye.1998.143. ISSN 0950-222X. PMID: 9775215.
- ↑ T. Ryan Gregory, Paul D.N. Hebert. The Modulation of DNA Content: Proximate Causes and Ultimate Consequences. „Genome Research”. 9 (4), s. 317–324, kwiecień 1999. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. DOI: 10.1101/gr.9.4.317. ISSN 1088-9051. PMID: 10207154. [dostęp 2014-12-11].
- ↑ Matthew Hurles. Gene Duplication: The Genomic Trade in Spare Parts. „PLOS Biology”. 2 (7), s. e206, 2004-07-13. San Francisco, CA: Public Library of Science. DOI: 10.1371/journal.pbio.0020206. ISSN 1545-7885. PMID: 15252449. PMCID: PMC449868.
- ↑ Na Liu, Katsutomo Okamura, David M. Tyler, Michael D. Phillips i inni. The evolution and functional diversification of animal microRNA genes. „Cell Research”. 18 (10), s. 985–996, 2008. London: Nature Publishing Group on behalf of the Shanghai Institutes for Biological Sciences. DOI: 10.1038/cr.2008.278. ISSN 1001-0602. PMID: 18711447. PMCID: PMC2712117. [dostęp 2014-12-11].
- ↑ Adam Siepel. Darwinian alchemy: Human genes from noncoding DNA. „Genome Research”. 19 (10), s. 1693–1695, październik 2009. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. DOI: 10.1101/gr.098376.109. ISSN 1088-9051. PMID: 19797681. PMCID: PMC2765273. [dostęp 2014-12-11].
- ↑ Christine A. Orengo, Janet M. Thornton. Protein families and their evolution–a structural perspective. „Annual Review of Biochemistry”. 74, s. 867–900, 2005. Palo Alto, CA: Annual Reviews. DOI: 10.1146/annurev.biochem.74.082803.133029. ISSN 0066-4154. PMID: 15954844.
- ↑ Manyuan Long, Esther Betrán, Kevin Thornton, Wen Wang. The origin of new genes: glimpses from the young and old. „Nature Reviews Genetics”. 4 (11), s. 865–875, listopad 2003. London: Nature Publishing Group. DOI: 10.1038/nrg1204. ISSN 1471-0056. PMID: 14634634.
- ↑ Minglei Wang, Gustavo Caetano-Anollés. The Evolutionary Mechanics of Domain Organization in Proteomes and the Rise of Modularity in the Protein World. „Structure”. 17 (1), s. 66–78, 2009-01-14. Cambridge, MA: Cell Press. DOI: s. 2008.11.008 10.1016/j. s. 2008.11.008. ISSN 1357-4310. PMID: 19141283.
- ↑ Kira J. Weissman, Rolf Müller. Protein–Protein Interactions in Multienzyme Megasynthetases. „ChemBioChem”. 9 (6), s. 826–848, 2008-04-14. Weinheim, Germany: Wiley-VCH. DOI: 10.1002/cbic.200700751. ISSN 1439-4227. PMID: 18357594.
- ↑ Charles M. Radding. Homologous Pairing and Strand Exchange in Genetic Recombination. „Annual Review of Genetics”. 16, s. 405–437, 1982. Palo Alto, CA: Annual Reviews. DOI: 10.1146/annurev.ge.16.120182.002201. ISSN 0066-4197. PMID: 6297377.
- ↑ Aneil F. Agrawal. Evolution of Sex: Why Do Organisms Shuffle Their Genotypes?. „Current Biology”. 16 (17), s. R696–R704, 2006-09-05. Cambridge, MA: Cell Press. DOI: 10.1016/j.cub.2006.07.063. ISSN 0960-9822. PMID: 16950096.
- ↑ Andrew D. Peters, Sarah P. Otto. Liberating genetic variance through sex. „BioEssays”. 25 (6), s. 533–537, 2003. Hoboken, NJ: John Wiley & Sons. DOI: 10.1002/bies.10291. ISSN 0265-9247. PMID: 12766942.
- ↑ Matthew R. Goddard, H. Charles J. Godfray, Austin Burt. Sex increases the efficacy of natural selection in experimental yeast populations. „Nature”. 434 (7033), s. 636–640, 2005-03-31. London: Nature Publishing Group. DOI: 10.1038/nature03405. ISSN 0028-0836. PMID: 15800622. Bibcode: 2005Natur.434..636G.
- ↑ Maynard Smith, 1978.
- ↑ a b Ridley 1993 ↓.
- ↑ Leigh Van Valen. A New Evolutionary Law. „Evolutionary Theory”. 1, s. 1–30, 1973. Chicago, IL: University of Chicago. ISSN 0093-4755. [dostęp 2014-12-24]. [zarchiwizowane z adresu].
- ↑ W.D. Hamilton, Robert Axelrod, Reiko Tanese. Sexual reproduction as an adaptation to resist parasites (a review). „Proc. Natl. Acad. Sci. U.S.A.”. 87 (9), s. 3566–3573, 1990-05-01. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.87.9.3566. ISSN 0027-8424. PMID: 2185476. Bibcode: 1990PNAS...87.3566H.
- ↑ Birdsell i Wills 2003 ↓, s. 113–117.
- ↑ a b Carrie L. Morjan, Loren H. Rieseberg. How species evolve collectively: implications of gene flow and selection for the spread of advantageous alleles. „Molecular Ecology”. 13 (6), s. 1341–1356, 2004. Hoboken, NJ: Wiley-Blackwell. DOI: 10.1111/j.1365-294X.2004.02164.x. ISSN 0962-1083. PMID: 15140081. PMCID: PMC2600545.
- ↑ Tarchand, Johry, Saxena & Kathpolia: General Science for Civil Service Preliminary Examination Paper 1. Tata McGraw-Hill Education, s. B. 154. ISBN 978-1-259-00121-5.
- ↑ Yan Boucher, Christophe J. Douady, R. Thane Papke, David A. Walsh i inni. Lateral gene transfer and the origins of prokaryotic groups. „Annual Review of Genetics”. 37, s. 283–328, 2003. Palo Alto, CA: Annual Reviews. DOI: 10.1146/annurev.genet.37.050503.084247. ISSN 0066-4197. PMID: 14616063.
- ↑ Timothy R. Walsh. Combinatorial genetic evolution of multiresistance. „Current Opinion in Microbiology”. 9 (5), s. 476–482, październik 2006. Amsterdam, the Netherlands: Elsevier. DOI: 10.1016/j.mib.2006.08.009. ISSN 1369-5274. PMID: 16942901.
- ↑ Natsuko Kondo, Naruo Nikoh, Nobuyuki Ijichi, Masakazu Shimada i inni. Genome fragment of Wolbachia endosymbiont transferred to X chromosome of host insect. „Proc. Natl. Acad. Sci. U.S.A.”. 99 (22), s. 14280–14285, 2002-10-29. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.222228199. ISSN 0027-8424. PMID: 12386340. PMCID: PMC137875. Bibcode: 2002PNAS...9914280K.
- ↑ George F., Jr. Sprague. Genetic exchange between kingdoms. „Current Opinion in Genetics & Development”. 1 (4), s. 530–533, 1991. Amsterdam, the Netherlands: Elsevier. DOI: 10.1016/S0959-437X(05)80203-5. ISSN 0959-437X. PMID: 1822285.
- ↑ Eugene A. Gladyshev, Matthew Meselson, Irina R. Arkhipova. Massive Horizontal Gene Transfer in Bdelloid Rotifers. „Science”. 320 (5880), s. 1210–1213, 2008-05-30. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.1156407. ISSN 0036-8075. PMID: 18511688. Bibcode: 2008Sci...320.1210G.
- ↑ Angela M. Baldo, Marcella A. McClure. Evolution and Horizontal Transfer of dUTPase-Encoding Genes in Viruses and Their Hosts. „Journal of Virology”. 73 (9), s. 7710–7721, 1999. Washington, D.C.: American Society for Microbiology. ISSN 0022-538X. PMID: 10438861. PMCID: PMC104298.
- ↑ Maria C. Rivera, James A. Lake. The ring of life provides evidence for a genome fusion origin of eukaryotes. „Nature”. 431 (7005), s. 152–155, 2004-09-09. London: Nature Publishing Group. DOI: 10.1038/nature02848. ISSN 0028-0836. PMID: 15356622. Bibcode: 2004Natur.431..152R.
- ↑ a b c Laurence D. Hurst. Fundamental concepts in genetics: genetics and the understanding of selection. „Nature Reviews Genetics”. 10 (2), s. 83–93, luty 2009. London: Nature Publishing Group. DOI: 10.1038/nrg2506. ISSN 1471-0056. PMID: 19119264.
- ↑ a b c H. Allen Orr. Fitness and its role in evolutionary genetics. „Nature Reviews Genetics”. 10 (8), s. 531–539, 2009. London: Nature Publishing Group. DOI: 10.1038/nrg2603. ISSN 1471-0056. PMID: 19546856. PMCID: PMC2753274.
- ↑ J.B.S. Haldane. The Theory of Natural Selection To-Day. „Nature”. 183 (4663), s. 710–713, 14 marca 1959. London: Nature Publishing Group. DOI: 10.1038/183710a0. ISSN 0028-0836. PMID: 13644170. Bibcode: 1959Natur.183..710H.
- ↑ Russell Lande, Stevan J. Arnold. The Measurement of Selection on Correlated Characters. „Evolution”. 37 (6), s. 1210–1226, 1983. Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution. DOI: 10.2307/2408842. ISSN 0014-3820. JSTOR: 2408842.
- ↑ Emma E. Goldberg, Boris Igić. On phylogenetic tests of irreversible evolution. „Evolution”. 62 (11), s. 2727–2741, 2008. Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution. DOI: 10.1111/j.1558-5646.2008.00505.x. ISSN 0014-3820. PMID: 18764918.
- ↑ Rachel Collin, Maria Pia Miglietta. Reversing opinions on Dollo’s Law. „Trends in Ecology & Evolution”. 23 (11), s. 602–609, listopad 2008. Cambridge, MA: Cell Press. DOI: 10.1016/j.tree.2008.06.013. ISSN 0169-5347. PMID: 18814933.
- ↑ Hopi E. Hoekstra, Jonathan M. Hoekstra, David Berrigan, Sacha N. Vignieri i inni. Strength and tempo of directional selection in the wild. „Proc. Natl. Acad. Sci. U.S.A.”. 98 (16), s. 9157–9160, 2001-07-31. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.161281098. ISSN 0027-8424. PMID: 11470913. PMCID: PMC55389. Bibcode: 2001PNAS...98.9157H.
- ↑ Joseph Felsenstein. Excursions along the Interface between Disruptive and Stabilizing Selection. „Genetics”. 93 (3), s. 773–795, 1979. Bethesda, MD: Genetics Society of America. ISSN 0016-6731. PMID: 17248980. PMCID: PMC1214112.
- ↑ Malte Andersson, Leigh W. Simmons. Sexual selection and mate choice. „Trends in Ecology & Evolution”. 21 (6), s. 296–302, 2006. Cambridge, MA: Cell Press. DOI: 10.1016/j.tree.2006.03.015. ISSN 0169-5347. PMID: 16769428.
- ↑ Hanna Kokko, Robert Brooks, John M. McNamara, Alasdair I. Houston. The sexual selection continuum. „Proceedings of the Royal Society B”. 269 (1498), s. 1331–1340, 2002-07-07. London: Royal Society. DOI: 10.1098/rspb.2002.2020. ISSN 0962-8452. PMID: 12079655. PMCID: PMC1691039.
- ↑ Thomas P. Quinn, Andrew P. Hendry, Gregory B. Buck. Balancing natural and sexual selection in sockeye salmon: interactions between body size, reproductive opportunity and vulnerability to predation by bears. „Evolutionary Ecology Research”. 3, s. 917–937, 2001. ISSN 1522-0613. [dostęp 2014-12-15].
- ↑ John Hunt, Robert Brooks, Michael D. Jennions, Michael J. Smith i inni. High-quality male field crickets invest heavily in sexual display but die young. „Nature”. 432 (7020), s. 1024–1027, 2004-12-23. London: Nature Publishing Group. DOI: 10.1038/nature03084. ISSN 0028-0836. PMID: 15616562. Bibcode: 2004Natur.432.1024H.
- ↑ Odum 1971 ↓, s. 8.
- ↑ Okasha 2006 ↓.
- ↑ a b Stephen Jay Gould. Gulliver’s further travels: the necessity and difficulty of a hierarchical theory of selection. „Philosophical Transactions of the Royal Society B: Biological Sciences”. 353 (1366), s. 307–314, 28 lutego 1998. London: Royal Society. DOI: 10.1098/rstb.1998.0211. ISSN 0962-8436. PMID: 9533127. PMCID: PMC1692213.
- ↑ Ernst Mayr. The objects of selection. „Proc. Natl. Acad. Sci. U.S.A.”. 94 (6), s. 2091–2094, 18 marca 1997. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.94.6.2091. ISSN 0027-8424. PMID: 9122151. PMCID: PMC33654. Bibcode: 1997PNAS...94.2091M.
- ↑ Maynard Smith 1998 ↓, s. 203–217.
- ↑ Donal A. Hickey. Evolutionary dynamics of transposable elements in prokaryotes and eukaryotes. „Genetica”. 86 (1–3), s. 269–274, 1992. Dordrecht, the Netherlands: Kluwer Academic Publishers. DOI: 10.1007/BF00133725. ISSN 0016-6707. PMID: 1334911.
- ↑ Stephen Jay Gould, Elisabeth A. Lloyd. Individuality and adaptation across levels of selection: how shall we name and generalise the unit of Darwinism?. „Proc. Natl. Acad. Sci. U.S.A.”. 96 (21), s. 11904–11909, 1999-10-12. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.96.21.11904. ISSN 0027-8424. PMID: 10518549. PMCID: PMC18385. Bibcode: 1999PNAS...9611904G.
- ↑ Michael Lynch. The frailty of adaptive hypotheses for the origins of organismal complexity. „Proc. Natl. Acad. Sci. U.S.A.”. 104 (Suppl. 1), s. 8597–8604, 2007-05-15. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0702207104. ISSN 0027-8424. PMID: 17494740. PMCID: PMC1876435. Bibcode: 2007PNAS..104.8597L.
- ↑ Nick G.C. Smith, Matthew T. Webster, Hans Ellegren. Deterministic Mutation Rate Variation in the Human Genome. „Genome Research”. 12 (9), s. 1350–1356, 2002. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. DOI: 10.1101/gr.220502. ISSN 1088-9051. PMID: 12213772. PMCID: PMC186654.
- ↑ Dmitri A. Petrov, Todd A. Sangster, J. Spencer Johnston, Daniel L. Hartl i inni. Evidence for DNA Loss as a Determinant of Genome Size. „Science”. 287 (5455), s. 1060–1062, 11 lutego 2000. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.287.5455.1060. ISSN 0036-8075. PMID: 10669421. Bibcode: 2000Sci...287.1060P.
- ↑ Dmitri A. Petrov. DNA loss and evolution of genome size in Drosophila. „Genetica”. 115 (1), s. 81–91, maj 2002. Dordrecht, the Netherlands: Kluwer Academic Publishers. DOI: 10.1023/A:1016076215168. ISSN 0016-6707. PMID: 12188050.
- ↑ Karin Kiontke, Antoine Barriere, Irina Kolotuev, Benjamin Podbilewicz i inni. Trends, Stasis, and Drift in the Evolution of Nematode Vulva Development. „Current Biology”. 17 (22), s. 1925–1937, listopad 2007. Cambridge, MA: Cell Press. DOI: 10.1016/j.cub.2007.10.061. ISSN 0960-9822. PMID: 18024125.
- ↑ Christian Braendle, Charles F. Baer, Marie-Anne Félix, Gregory S. Barsh (red.). Bias and Evolution of the Mutationally Accessible Phenotypic Space in a Developmental System. „PLOS Genetics”. 6 (3), s. e1000877, 2010-03-12. San Francisco, CA: Public Library of Science. DOI: 10.1371/journal.pgen.1000877. ISSN 1553-7390. PMID: 20300655. PMCID: PMC2837400.
- ↑ A. Richard Palmer. Symmetry breaking and the evolution of development. „Science”. 306 (5697), s. 828–833, 2004-10-29. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.1103707. ISSN 0036-8075. PMID: 15514148. Bibcode: 2004Sci...306..828P.
- ↑ West-Eberhard 2003 ↓.
- ↑ Arlin Stoltzfus, Lev Y. Yampolsky. Climbing Mount Probable: Mutation as a Cause of Nonrandomness in Evolution. „Journal of Heredity”. 100 (5), s. 637–647, wrzesień–październik 2009. Oxford: Oxford University Press on behalf of the American Genetic Association. DOI: 10.1093/jhered/esp048. ISSN 0022-1503. PMID: 19625453.
- ↑ Lev Y. Yampolsky, Arlin Stoltzfus. Bias in the introduction of variation as an orienting factor in evolution. „Evolution & Development”. 3 (2), s. 73–83, marzec 2001. Hoboken, NJ: Wiley-Blackwell on behalf of the Society for Integrative and Comparative Biology. DOI: 10.1046/j.1525-142x.2001.003002073.x. ISSN 1520-541X. PMID: 11341676.
- ↑ J.B.S. Haldane. The Part Played by Recurrent Mutation in Evolution. „The American Naturalist”. 67 (708), s. 5–19, 1933. Chicago, IL: University of Chicago Press on behalf of the American Society of Naturalists. DOI: 10.1086/280465. ISSN 0003-0147. JSTOR: 2457127.
- ↑ Meredith Protas, Melissa Conrad, Joshua B. Gross, Clifford Tabin i inni. Regressive Evolution in the Mexican Cave Tetra, Astyanax mexicanus. „Current Biology”. 17 (5), s. 452–454, 2007-03-06. Cambridge, MA: Cell Press. DOI: 10.1016/j.cub.2007.01.051. ISSN 0960-9822. PMID: 17306543. PMCID: PMC2570642.
- ↑ Heather Maughan, Joanna Masel, C. William, Jr. Birky, Wayne L. Nicholson. The Roles of Mutation Accumulation and Selection in Loss of Sporulation in Experimental Populations of Bacillus subtilis. „Genetics”. 177 (2), s. 937–948, październik 2007. Bethesda, MD: Genetics Society of America. DOI: 10.1534/genetics.107.075663. ISSN 0016-6731. PMID: 17720926. PMCID: PMC2034656.
- ↑ Joanna Masel, Oliver D. King, Heather Maughan. The Loss of Adaptive Plasticity during Long Periods of Environmental Stasis. „The American Naturalist”. 169 (1), s. 38–46, 2007. Chicago, IL: University of Chicago Press on behalf of the American Society of Naturalists. DOI: 10.1086/510212. ISSN 0003-0147. PMID: 17206583. PMCID: PMC1766558.
- ↑ a b Joanna Masel. Genetic drift. „Current Biology”. 21 (20), s. R837–R838, 2011-10-25. Cambridge, MA: Cell Press. DOI: 10.1016/j.cub.2011.08.007. ISSN 0960-9822. PMID: 22032182.
- ↑ Russell Lande. Fisherian and Wrightian theories of speciation. „Genome”. 31 (1), s. 221–227, 1989. Ottawa: National Research Council of Canada. DOI: 10.1139/g89-037. ISSN 0831-2796. PMID: 2687093.
- ↑ Thomas Mitchell-Olds, John H. Willis, David B. Goldstein. Which evolutionary processes influence natural genetic variation for phenotypic traits?. „Nature Reviews Genetics”. 8 (11), s. 845–856, 2007. London: Nature Publishing Group. DOI: 10.1038/nrg2207. ISSN 1471-0056. PMID: 17943192.
- ↑ Masatoshi Nei. Selectionism and Neutralism in Molecular Evolution. „Molecular Biology and Evolution”. 22 (12), s. 2318–2342, grudzień 2005. Oxford: Oxford University Press on behalf of the Society for Molecular Biology and Evolution. DOI: 10.1093/molbev/msi242. ISSN 0737-4038. PMID: 16120807. PMCID: PMC1513187. Masatoshi Nei. Selectionism and Neutralism in Molecular Evolution. „Molecular Biology and Evolution”. 23 (5), s. 1095, 2006. Oxford: Oxford University Press on behalf of the Society for Molecular Biology and Evolution. DOI: 10.1093/molbev/msk009. ISSN 0737-4038.
- ↑ Motoo Kimura. The neutral theory of molecular evolution: a review of recent evidence. „The Japanese Journal of Human Genetics”. 66 (4), s. 367–386, 1991. Mishima, Japan: Genetics Society of Japan. DOI: 10.1266/jjg.66.367. ISSN 0021-504X. PMID: 1954033.
- ↑ Motoo Kimura. The neutral theory of molecular evolution and the world view of the neutralists. „Genome”. 31 (1), s. 24–31, 1989. Ottawa: National Research Council of Canada. DOI: 10.1139/g89-009. ISSN 0831-2796. PMID: 2687096.
- ↑ Martin Kreitman. The neutral theory is dead. Long live the neutral theory. „BioEssays”. 18 (8), s. 678–683; discussion 683, sierpień 1996. Hoboken, NJ: John Wiley & Sons. DOI: 10.1002/bies.950180812. ISSN 0265-9247. PMID: 8760341.
- ↑ E. G., Jr. Leigh. Neutral theory: a historical perspective. „Journal of Evolutionary Biology”. 20 (6), s. 2075–2091, listopad 2007. Hoboken, NJ: Wiley-Blackwell on behalf of the European Society for Evolutionary Biology. DOI: 10.1111/j.1420-9101.2007.01410.x. ISSN 1010-061X. PMID: 17956380.
- ↑ a b John H. Gillespie. Is the population size of a species relevant to its evolution?. „Evolution”. 55 (11), s. 2161–2169, 2001. Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution. DOI: 10.1111/j.0014-3820.2001.tb00732.x. ISSN 0014-3820. PMID: 11794777.
- ↑ Richard A. Neher, Boris I. Shraiman. Genetic Draft and Quasi-Neutrality in Large Facultatively Sexual Populations. „Genetics”. 188 (4), s. 975–996, sierpień 2011. Bethesda, MD: Genetics Society of America. DOI: 10.1534/genetics.111.128876. ISSN 0016-6731. PMID: 21625002. PMCID: PMC3176096.
- ↑ Sarah P. Otto, Michael C. Whitlock. The Probability of Fixation in Populations of Changing Size. „Genetics”. 146 (2), s. 723–733, czerwiec 1997. Bethesda, MD: Genetics Society of America. ISSN 0016-6731. PMID: 9178020. PMCID: PMC1208011. [dostęp 2014-12-18].
- ↑ a b Brian Charlesworth. Fundamental concepts in genetics: effective population size and patterns of molecular evolution and variation. „Nature Reviews Genetics”. 10 (3), s. 195–205, marzec 2009. London: Nature Publishing Group. DOI: 10.1038/nrg2526. ISSN 1471-0056. PMID: 19204717.
- ↑ Asher D. Cutter, Jae Young Choi. Natural selection shapes nucleotide polymorphism across the genome of the nematode Caenorhabditis briggsae. „Genome Research”. 20 (8), s. 1103–1111, 2010. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. DOI: 10.1101/gr.104331.109. ISSN 1088-9051. PMID: 20508143. PMCID: PMC2909573.
- ↑ Sigbjørn Lien, Joanna Szyda, Birgit Schechinger, Gudrun Rappold i inni. Evidence for Heterogeneity in Recombination in the Human Pseudoautosomal Region: High Resolution Analysis by Sperm Typing and Radiation-Hybrid Mapping. „American Journal of Human Genetics”. 66 (2), s. 557–566, luty 2000. Cambridge, MA: Cell Press on behalf of the American Society of Human Genetics. DOI: 10.1086/302754. ISSN 0002-9297. PMID: 10677316. PMCID: PMC1288109.
- ↑ Nicholas H. Barton. Genetic hitchhiking. „Philosophical Transactions of the Royal Society B: Biological Sciences”. 355 (1403), s. 1553–1562, 2000-11-29. London: Royal Society. DOI: 10.1098/rstb.2000.0716. ISSN 0962-8436. PMID: 11127900. PMCID: PMC1692896.
- ↑ Futuyma 2008 ↓, s. 218.
- ↑ Sewall Wright. The roles of mutation, inbreeding, crossbreeding and selection in evolution. „Proceedings of the VI International Congress of Genetrics”. 1, s. 356–366, 1932. [dostęp 2014-12-18].
- ↑ Jerry A. Coyne, Nicholas H. Barton, Michael Turelli. Perspective: A Critique of Sewall Wright’s Shifting Balance Theory of Evolution. „Evolution”. 51 (3), s. 643–671, 1997. Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution. DOI: 10.2307/2411143. ISSN 0014-3820.
- ↑ Futuyma 2008 ↓, s. 507.
- ↑ a b c d Eugenie C. Scott, Nicholas J. Matzke. Biological design in science classrooms. „Proc. Natl. Acad. Sci. U.S.A.”. 104 (Suppl. 1), s. 8669–8676, 2007-05-15. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0701505104. ISSN 0027-8424. PMID: 17494747. PMCID: PMC1876445. Bibcode: 2007PNAS..104.8669S.
- ↑ Andrew Paul Hendry, Michael T. Kinnison. An introduction to microevolution: rate, pattern, process. „Genetica”. 112–113 (1), s. 1–8, listopad 2001. Dordrecht, the Netherlands: Kluwer Academic Publishers. DOI: 10.1023/A:1013368628607. ISSN 0016-6707. PMID: 11838760.
- ↑ Armand M. Leroi. The scale independence of evolution. „Evolution & Development”. 2 (2), s. 67–77, marzec–kwiecień 2000. Hoboken, NJ: Wiley-Blackwell on behalf of the Society for Integrative and Comparative Biology. DOI: 10.1046/j.1525-142x.2000.00044.x. ISSN 1520-541X. PMID: 11258392.
- ↑ Gould 2002 ↓, s. 657–658.
- ↑ a b Stephen Jay Gould. Tempo and mode in the macroevolutionary reconstruction of Darwinism. „Proc. Natl. Acad. Sci. U.S.A.”. 91 (15), s. 6764–6771, 19 lipca 1994. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.91.15.6764. ISSN 0027-8424. PMID: 8041695. PMCID: PMC44281. Bibcode: 1994PNAS...91.6764G.
- ↑ David Jablonski, Micro- and macroevolution: scale and hierarchy in evolutionary biology and paleobiology, „Paleobiology”, sp4, 26, Boulder, CO: Paleontological Society, 2000, s. 15–52, DOI: 10.1666/0094-8373(2000)26[15:MAMSAH]2.0.CO;2, ISSN 0094-8373.
- ↑ Sean B. Carroll. Chance and necessity: the evolution of morphological complexity and diversity. „Nature”. 409 (6823), s. 1102–1109, 22 lutego 2001. London: Nature Publishing Group. DOI: 10.1038/35059227. ISSN 0028-0836. PMID: 11234024. Bibcode: 2001Natur.409.1102C.
- ↑ William B. Whitman, David C. Coleman, William J. Wiebe. Prokaryotes: The unseen majority. „Proc. Natl. Acad. Sci. U.S.A.”. 95 (12), s. 6578–6583, 1998-06-09. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.95.12.6578. ISSN 0027-8424. PMID: 9618454. PMCID: PMC33863. Bibcode: 1998PNAS...95.6578W.
- ↑ a b Patrick D. Schloss, Jo Handelsman. Status of the Microbial Census. „Microbiology and Molecular Biology Reviews”. 68 (4), s. 686–691, 2004. Washington, D.C.: American Society for Microbiology. DOI: 10.1128/MMBR.68.4.686-691.2004. ISSN 1092-2172. PMID: 15590780. PMCID: PMC539005.
- ↑ Kenneth H. Nealson. Post-Viking microbiology: new approaches, new data, new insights. „Origins of Life and Evolution of Biospheres”. 29 (1), s. 73–93, styczeń 1999. Dordrecht, the Netherlands: Kluwer Academic Publishers. DOI: 10.1023/A:1006515817767. ISSN 0169-6149. PMID: 11536899.
- ↑ Angus Buckling, R. Craig MacLean, Michael A. Brockhurst, Nick Colegrave. The Beagle in a bottle. „Nature”. 457 (7231), s. 824–829, 2009-02-12. London: Nature Publishing Group. DOI: 10.1038/nature07892. ISSN 0028-0836. PMID: 19212400. Bibcode: 2009Natur.457..824B.
- ↑ Santiago F. Elena, Richard E. Lenski. Evolution experiments with microorganisms: the dynamics and genetic bases of adaptation. „Nature Reviews Genetics”. 4 (6), s. 457–469, 2003. London: Nature Publishing Group. DOI: 10.1038/nrg1088. ISSN 1471-0056. PMID: 12776215.
- ↑ Mayr 1982 ↓, s. 483.
- ↑ Oxford Dictionary of Science, wydanie 6 (2010).
- ↑ H. Allen Orr. The genetic theory of adaptation: a brief history. „Nature Reviews Genetics”. 6 (2), s. 119–127, 2005. London: Nature Publishing Group. DOI: 10.1038/nrg1523. ISSN 1471-0056. PMID: 15716908.
- ↑ Dobzhansky 1968 ↓, s. 1–34.
- ↑ Dobzhansky 1970 ↓, s. 4–6, 79–82, 84–87.
- ↑ Theodosius Dobzhansky. Genetics of Natural Populations. XXV. Genetic Changes in Populations of Drosophila pseudoobscura and Drosophila persimilis in Some Localities in California. „Evolution”. 10 (1), s. 82–92, 1956. Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution. DOI: 10.2307/2406099. ISSN 0014-3820. JSTOR: 2406099.
- ↑ Akira Nakajima, Yohko Sugimoto, Hiroshi Yoneyama, Taiji Nakae. High-Level Fluoroquinolone Resistance in Pseudomonas aeruginosa Due to Interplay of the MexAB-OprM Efflux Pump and the DNA Gyrase Mutation. „Microbiology and Immunology”. 46 (6), s. 391–395, czerwiec 2002. Tokyo: Center for Academic Publications Japan. DOI: 10.1111/j.1348-0421.2002.tb02711.x. ISSN 1348-0421. PMID: 12153116.
- ↑ Zachary D. Blount, Christina Z. Borland, Richard E. Lenski. Inaugural Article: Historical contingency and the evolution of a key innovation in an experimental population of Escherichia coli. „Proc. Natl. Acad. Sci. U.S.A.”. 105 (23), s. 7899–7906, 2008-06-10. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0803151105. ISSN 0027-8424. PMID: 18524956. PMCID: PMC2430337. Bibcode: 2008PNAS..105.7899B.
- ↑ Hirosuke Okada, Seiji Negoro, Hiroyuki Kimura, Shunichi Nakamura. Evolutionary adaptation of plasmid-encoded enzymes for degrading nylon oligomers. „Nature”. 306 (5939), s. 203–206, 1983-11-10. London: Nature Publishing Group. DOI: 10.1038/306203a0. ISSN 0028-0836. PMID: 6646204. Bibcode: 1983Natur.306..203O.
- ↑ Susumu Ohno. Birth of a unique enzyme from an alternative reading frame of the preexisted, internally repetitious coding sequence. „Proc. Natl. Acad. Sci. U.S.A.”. 81 (8), s. 2421–2425, 1984. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.81.8.2421. ISSN 0027-8424. PMID: 6585807. PMCID: PMC345072. Bibcode: 1984PNAS...81.2421O.
- ↑ Shelley D. Copley. Evolution of a metabolic pathway for degradation of a toxic xenobiotic: the patchwork approach. „Trends in Biochemical Sciences”. 25 (6), s. 261–265, 2000. Cambridge, MA: Cell Press. DOI: 10.1016/S0968-0004(00)01562-0. ISSN 0968-0004. PMID: 10838562.
- ↑ Ronald L. Crawford, Carina M. Jung, Janice L. Strap. The recent evolution of pentachlorophenol (PCP)-4-monooxygenase (PcpB) and associated pathways for bacterial degradation of PCP. „Biodegradation”. 18 (5), s. 525–539, 2007. Dordrecht, the Netherlands: Springer Netherlands. DOI: 10.1007/s10532-006-9090-6. ISSN 0923-9820. PMID: 17123025.
- ↑ Ilan Eshel. Clone-Selection and Optimal Rates of Mutation. „Journal of Applied Probability”. 10 (4), s. 728–738, 1973. Sheffield, England. DOI: 10.2307/3212376. ISSN 1475-6072. JSTOR: 3212376.
- ↑ Altenberg 1995 ↓, s. 205–259.
- ↑ Joanna Masel, Aviv Bergman. The evolution of the evolvability properties of the yeast prion [PSI+]. „Evolution”. 57 (7), s. 1498–1512, 2003. Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution. DOI: 10.1111/j.0014-3820.2003.tb00358.x. ISSN 0014-3820. PMID: 12940355.
- ↑ Alex K. Lancaster, J. Patrick Bardill, Heather L. True, Joanna Masel. The Spontaneous Appearance Rate of the Yeast Prion [PSI+] and Its Implications for the Evolution of the Evolvability Properties of the [PSI+] System. „Genetics”. 184 (2), s. 393–400, 2010. Bethesda, MD: Genetics Society of America. DOI: 10.1534/genetics.109.110213. ISSN 0016-6731. PMID: 19917766. PMCID: PMC2828720.
- ↑ Jeremy Draghi, Günter P. Wagner. Evolution of evolvability in a developmental model. „Evolution”. 62 (2), s. 301–315, 2008. Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution. DOI: 10.1111/j.1558-5646.2007.00303.x. ISSN 0014-3820. PMID: 18031304.
- ↑ a b Lars Bejder, Brian K. Hall. Limbs in whales and limblessness in other vertebrates: mechanisms of evolutionary and developmental transformation and loss. „Evolution & Development”. 4 (6), s. 445–458, 2002. Hoboken, NJ: Wiley-Blackwell on behalf of the Society for Integrative and Comparative Biology. DOI: 10.1046/j.1525-142X.2002.02033.x. ISSN 1520-541X. PMID: 12492145.
- ↑ Nathan M. Young, Benedikt HallgrÍmsson. Serial homology and the evolution of mammalian limb covariation structure. „Evolution”. 59 (12), s. 2691–2704, grudzień 2005. Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution. DOI: 10.1554/05-233.1. ISSN 0014-3820. PMID: 16526515.
- ↑ a b David Penny, Anthony Poole. The nature of the last universal common ancestor. „Current Opinion in Genetics & Development”. 9 (6), s. 672–677, 1999. Amsterdam, Holandia: Elsevier. DOI: 10.1016/S0959-437X(99)00020-9. ISSN 0959-437X. PMID: 10607605.
- ↑ Brian K. Hall. Descent with modification: the unity underlying homology and homoplasy as seen through an analysis of development and evolution. „Biological Reviews”. 78 (3), s. 409–433, 2003. Cambridge: Cambridge University Press on behalf of the Cambridge Philosophical Society. DOI: 10.1017/S1464793102006097. ISSN 1464-7931. PMID: 14558591.
- ↑ Neil Shubin, Clifford J. Tabin, Sean Carroll. Deep homology and the origins of evolutionary novelty. „Nature”. 457 (7231), s. 818–823, 2009-02-12. London: Nature Publishing Group. DOI: 10.1038/nature07891. ISSN 0028-0836. PMID: 19212399. Bibcode: 2009Natur.457..818S.
- ↑ a b c Daniel F. Fong, Thomas C. Kane, David C. Culver. Vestigialization and Loss of Nonfunctional Characters. „Annual Review of Ecology and Systematics”. 26, s. 249–268, 1995. Palo Alto, CA: Annual Reviews. DOI: 10.1146/annurev.es.26.110195.001341. ISSN 1545-2069.
- ↑ ZhaoLei Zhang, Mark Gerstein. Large-scale analysis of pseudogenes in the human genome. „Current Opinion in Genetics & Development”. 14 (4), s. 328–335, 2004. Amsterdam, the Netherlands: Elsevier. DOI: 10.1016/j.gde.2004.06.003. ISSN 0959-437X. PMID: 15261647.
- ↑ William R. Jeffery. Adaptive Evolution of Eye Degeneration in the Mexican Blind Cavefish. „Journal of Heredity”. 96 (3), s. 185–196, 2005. Oxford: Oxford University Press on behalf of the American Genetic Association. DOI: 10.1093/jhered/esi028. ISSN 0022-1503. PMID: 15653557.
- ↑ Erin E. Maxwell, Hans C.E. Larsson. Osteology and myology of the wing of the Emu (Dromaius novaehollandiae) and its bearing on the evolution of vestigial structures. „Journal of Morphology”. 268 (5), s. 423–441, maj 2007. Hoboken, NJ: John Wiley & Sons. DOI: 10.1002/jmor.10527. ISSN 0362-2525. PMID: 17390336.
- ↑ Casper J. van der Kooi, Tanja Schwander. On the fate of sexual traits under asexuality. „Biological Reviews”. 89 (4), s. 805–819, 2014. Cambridge: Cambridge University Press on behalf of the Cambridge Philosophical Society. DOI: 10.1111/brv.12078. ISSN 1464-7931. PMID: 24443922. [dostęp 2015-08-05].
- ↑ Anthony R., Jr. Silvestri, Iqbal Singh. The unresolved problem of the third molar: Would people be better off without it?. „Journal of the American Dental Association”. 134 (4), s. 450–455, kwiecień 2003. Chicago, IL: American Dental Association. DOI: 10.14219/jada.archive.2003.0194. ISSN 0002-8177. PMID: 12733778.
- ↑ Coyne 2009 ↓, s. 62.
- ↑ Darwin 1872 ↓, s. 101, 103.
- ↑ Gray 2007 ↓, s. 66.
- ↑ Coyne 2009 ↓, s. 85–86.
- ↑ Stevens 1982 ↓, s. 87.
- ↑ a b Gould 2002 ↓, s. 1235–1236.
- ↑ Mark J. Pallen, Nicholas J. Matzke. From The Origin of Species to the origin of bacterial flagella. „Nature Reviews Microbiology”. 4 (10), s. 784–790, 2006. London: Nature Publishing Group. DOI: 10.1038/nrmicro1493. ISSN 1740-1526. PMID: 16953248. [dostęp 2014-12-25]. [zarchiwizowane z adresu].
- ↑ Abigail Clements, Dejan Bursac, Xenia Gatsos, Andrew J. Perry i inni. The reducible complexity of a mitochondrial molecular machine. „Proc. Natl. Acad. Sci. U.S.A.”. 106 (37), s. 15791–15795, S 15-09-2009. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0908264106. ISSN 0027-8424. PMID: 19717453. PMCID: PMC2747197. Bibcode: 2009PNAS..10615791C.
- ↑ Piatigorsky i in. 1994 ↓, s. 241–250.
- ↑ Graeme Wistow. Lens crystallins: gene recruitment and evolutionary dynamism. „Trends in Biochemical Sciences”. 18 (8), s. 301–306, 1993. Cambridge, MA: Cell Press. DOI: 10.1016/0968-0004(93)90041-K. ISSN 0968-0004. PMID: 8236445.
- ↑ Norman A. Johnson, Adam H. Porter. Toward a new synthesis: population genetics and evolutionary developmental biology. „Genetica”. 112–113 (1), s. 45–58, listopad 2001. Dordrecht, Holandia: Kluwer Academic Publishers. DOI: 10.1023/A:1013371201773. ISSN 0016-6707. PMID: 11838782.
- ↑ Jaume Baguñà, Jordi Garcia-Fernàndez. Evo-Devo: the long and winding road. „The International Journal of Developmental Biology”. 47 (7–8), s. 705–713, 2003. Bilbao, Spain: University of the Basque Country Press. ISSN 0214-6282. PMID: 14756346.
- ↑ Alan C. Love. Evolutionary Morphology, Innovation and the Synthesis of Evolutionary and Developmental Biology. „Biology and Philosophy”. 18 (2), s. 309–345, 2003. Dordrecht, Holandia: Kluwer Academic Publishers. DOI: 10.1023/A:1023940220348. ISSN 0169-3867.
- ↑ Edgar F. Allin. Evolution of the mammalian middle ear. „Journal of Morphology”. 147 (4), s. 403–437, 1975. Hoboken, NJ: John Wiley & Sons. DOI: 10.1002/jmor.1051470404. ISSN 0362-2525. PMID: 1202224.
- ↑ Matthew P. Harris, Sean M. Hasso, Mark W.J. Ferguson, John F. Fallon. The Development of Archosaurian First-Generation Teeth in a Chicken Mutant. „Current Biology”. 16 (4), s. 371–377, 2006-02-21. Cambridge, MA: Cell Press. DOI: 10.1016/j.cub.2005.12.047. ISSN 0960-9822. PMID: 16488870.
- ↑ Sean B. Carroll. Evo-Devo and an Expanding Evolutionary Synthesis: A Genetic Theory of Morphological Evolution. „Cell”. 134 (1), s. 25–36, 11 lipca 2008. Cambridge, MA: Cell Press. DOI: 10.1016/j.cell.2008.06.030. ISSN 0092-8674. PMID: 18614008.
- ↑ Michael J. Wade. The co-evolutionary genetics of ecological communities. „Nature Reviews Genetics”. 8 (3), s. 185–195, 2007. London: Nature Publishing Group. DOI: 10.1038/nrg2031. ISSN 1471-0056. PMID: 17279094.
- ↑ Shana Geffeney, Edmund D., Jr. Brodie, Peter C. Ruben, Edmund D., III Brodie. Mechanisms of Adaptation in a Predator-Prey Arms Race: TTX-Resistant Sodium Channels. „Science”. 297 (5585), s. 1336–1339, 2002-08-23. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.1074310. ISSN 0036-8075. PMID: 12193784. Bibcode: 2002Sci...297.1336G.
- Edmund D. Brodie Jr. B.J. Ridenhour E.D. Brodie III, The evolutionary response of predators to dangerous prey: hotspots and coldspots in the geographic mosaic of coevolution between garter snakes and newts, „Evolution”, 56 (10), Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution, 2002, s. 2067–2082, DOI: 10.1554/0014-3820(2002)056[2067:teropt]2.0.co;2, ISSN 0014-3820, PMID: 12449493.
- Sean B. Carroll. Whatever Doesn’t Kill Some Animals Can Make Them Deadly. „The New York Times”, 2009-12-21. New York: The New York Times Company. ISSN 0362-4331. [dostęp 2014-12-26].
- ↑ Joel L. Sachs. Cooperation within and among species. „Journal of Evolutionary Biology”. 19 (5), s. 1415–1418, 1426–1436, 2006. Hoboken, NJ: Wiley-Blackwell on behalf of the European Society for Evolutionary Biology. DOI: 10.1111/j.1420-9101.2006.01152.x. ISSN 1010-061X. PMID: 16910971.
- ↑ Martin A. Nowak. Five Rules for the Evolution of Cooperation. „Science”. 314 (5805), s. 1560–1563, 2006-12-08. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.1133755. ISSN 0036-8075. PMID: 17158317. PMCID: PMC3279745. Bibcode: 2006Sci...314.1560N.
- ↑ Uta Paszkowski. Mutualism and parasitism: the yin and yang of plant symbioses. „Current Opinion in Plant Biology”. 9 (4), s. 364–370, sierpień 2006. Amsterdam, the Netherlands: Elsevier. DOI: 10.1016/j.pbi.2006.05.008. ISSN 1369-5266. PMID: 16713732.
- ↑ Bettina Hause, Thomas Fester. Molecular and cell biology of arbuscular mycorrhizal symbiosis. „Planta”. 221 (2), s. 184–196, 2005. Berlin: Springer-Verlag. DOI: 10.1007/s00425-004-1436-x. ISSN 0032-0935. PMID: 15871030.
- ↑ John S. Bertram. The molecular biology of cancer. „Molecular Aspects of Medicine”. 21 (6), s. 167–223, grudzień 2000. Amsterdam, Holandia: Elsevier, International Union of Biochemistry and Molecular Biology. DOI: 10.1016/S0098-2997(00)00007-8. ISSN 0098-2997. PMID: 11173079.
- ↑ H. Kern Reeve, Bert Hölldobler. The emergence of a superorganism through intergroup competition. „Proc. Natl. Acad. Sci. U.S.A.”. 104 (23), s. 9736–9740, 5 czerwca 2007. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0703466104. ISSN 0027-8424. PMID: 17517608. PMCID: PMC1887545. Bibcode: 2007PNAS..104.9736R.
- ↑ Robert Axelrod, W.D. Hamilton. The evolution of cooperation. „Science”. 211 (4489), s. 1390–1396, 1981-03-27. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.7466396. ISSN 0036-8075. PMID: 7466396. Bibcode: 1981Sci...211.1390A.
- ↑ Edward O. Wilson, Bert Hölldobler. Eusociality: Origin and consequences. „Proc. Natl. Acad. Sci. U.S.A.”. 102 (38), s. 13367–11371, 20 września 2005. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0505858102. ISSN 0027-8424. PMID: 16157878. PMCID: PMC1224642. Bibcode: 2005PNAS..10213367W.
- ↑ a b Sergey Gavrilets. Perspective: models of speciation: what have we learned in 40 years?. „Evolution”. 57 (10), s. 2197–2215, 2003. Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution. DOI: 10.1554/02-727. ISSN 0014-3820. PMID: 14628909.
- ↑ a b c Kevin de Queiroz. Ernst Mayr and the modern concept of species. „Proc. Natl. Acad. Sci. U.S.A.”. 102 (Suppl. 1), s. 6600–6607, 2005-05-03. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0502030102. ISSN 0027-8424. PMID: 15851674. PMCID: PMC1131873. Bibcode: 2005PNAS..102.6600D.
- ↑ a b Marc Ereshefsky. Elimisponative pluralism. „Philosophy of Science”. 59 (4), s. 671–690, 1992. Chicago, IL: University of Chicago Press. DOI: 10.1086/289701. ISSN 0031-8248. JSTOR: 188136.
- ↑ Mayr 1942 ↓, s. 120.
- ↑ Christophe Fraser, Eric J. Alm, Martin F. Polz, Brian G. Spratt i inni. The Bacterial Species Challenge: Making Sense of Genetic and Ecological Diversity. „Science”. 323 (5915), s. 741–746, 2009-02-06. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.1159388. ISSN 0036-8075. PMID: 19197054. Bibcode: 2009Sci...323..741F.
- ↑ Futuyma 2008 ↓, s. 363.
- ↑ Roger Valentine Short. The contribution of the mule to scientific thought. „Journal of Reproduction and Fertility. Supplement”, s. 359–364, 1975. Society for Reproduction and Fertility. ISSN 0449-3087. PMID: 1107543. OCLC 1639439.
- ↑ Briana L. Gross, Loren H. Rieseberg. The Ecological Genetics of Homoploid Hybrid Speciation. „Journal of Heredity”. 96 (3), s. 241–252, 2005. Oxford: Oxford University Press on behalf of the American Genetic Association. DOI: 10.1093/jhered/esi026. ISSN 0022-1503. PMID: 15618301. PMCID: PMC2517139.
- ↑ John M. Burke, Michael L. Arnold. Genetics and the fitness of hybrids. „Annual Review of Genetics”. 35, s. 31–52, 2001. Palo Alto, CA: Annual Reviews. DOI: 10.1146/annurev.genet.35.102401.085719. ISSN 0066-4197. PMID: 11700276.
- ↑ Robert C. Vrijenhoek. Polyploid Hybrids: Multiple Origins of a Treefrog Species. „Current Biology”. 16 (7), s. R245–R247, 2006-04-04. Cambridge, MA: Cell Press. DOI: 10.1016/j.cub.2006.03.005. ISSN 0960-9822. PMID: 16581499.
- ↑ William R. Rice, Ellen E. Hostert. Laboratory Experiments on Speciation: What Have We Learned in 40 Years?. „Evolution”. 47 (6), s. 1637–1653, 1993. Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution. DOI: 10.2307/2410209. ISSN 0014-3820.
- ↑ Chris D. Jiggins, Jon R. Bridle. Speciation in the apple maggot fly: a blend of vintages?. „Trends in Ecology & Evolution”. 19 (3), s. 111–114, 2004. Cambridge, MA: Cell Press. DOI: 10.1016/j.tree.2003.12.008. ISSN 0169-5347. PMID: 16701238.
- ↑ Joseph Boxhorn, Observed Instances of Speciation, Houston, TX: The TalkOrigins Foundation, Inc., 1 września 1995 [dostęp 2008-12-26].
- ↑ James R. Weinberg, Victoria R. Starczak, Daniele Jörg. Evidence for Rapid Speciation Following a Founder Event in the Laboratory. „Evolution”. 46 (4), s. 1214–1220, 1992. Hoboken, NJ: John Wiley & Sons on behalf of the Society for the Study of Evolution. DOI: 10.2307/2409766. ISSN 0014-3820. JSTOR: 2409766.
- ↑ Anthony Herrel, Katleen Huyghe, Bieke Vanhooydonck, Thierry Backeljau i inni. Rapid large-scale evolutionary divergence in morphology and performance associated with exploitation of a different dietary resource. „Proc. Natl. Acad. Sci. U.S.A.”. 105 (12), s. 4792–4795, 2008-03-25. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0711998105. ISSN 0027-8424. PMID: 18344323. PMCID: PMC2290806. Bibcode: 2008PNAS..105.4792H.
- ↑ Jonathan B. Losos, Kenneth I. Warhelt, Thomas W. Schoener. Adaptive differentiation following experimental island colonization in Anolis lizards. „Nature”. 387 (6628), s. 70–73, 1997-05-01. London: Nature Publishing Group. DOI: 10.1038/387070a0. ISSN 0028-0836. Bibcode: 1997Natur.387...70L.
- ↑ Conrad J. Hoskin, Megan Higgle, Keith R. McDonald, Craig Moritz. Reinforcement drives rapid allopatric speciation. „Nature”. 437 (7063), s. 1353–1356, 2005-10-27. London: Nature Publishing Group. DOI: 10.1038/nature04004. ISSN 0028-0836. PMID: 16251964. Bibcode: 2005Natur.437.1353H.
- ↑ Alan R. Templeton. The Theory of Speciation VIA the Founder Principle. „Genetics”. 94 (4), s. 1011–1038, 1980. Bethesda, MD: Genetics Society of America. ISSN 0016-6731. PMID: 6777243. PMCID: PMC1214177. [dostęp 2014-12-29].
- ↑ Janis Antonovics. Evolution in closely adjacent plant populations X: long-term persistence of prereproductive isolation at a mine boundary. „Heredity”. 97 (1), s. 33–37, 2006. London: Nature Publishing Group for The Genetics Society. DOI: 10.1038/sj.hdy.6800835. ISSN 0018-067X. PMID: 16639420. [dostęp 2014-12-29].
- ↑ Patrik Nosil, Bernard J. Crespi, Regine Gries, Gerhard Gries. Natural selection and divergence in mate preference during speciation. „Genetica”. 129 (3), s. 309–327, 2007. Dordrecht, the Netherlands: Kluwer Academic Publishers. DOI: 10.1007/s10709-006-0013-6. ISSN 0016-6707. PMID: 16900317.
- ↑ Vincent Savolainen, Marie-Charlotte Anstett, Christian Lexer, Ian Hutton i inni. Sympatric speciation in palms on an oceanic island. „Nature”. 441 (7090), s. 210–213, 2006-05-11. London: Nature Publishing Group. DOI: 10.1038/nature04566. ISSN 0028-0836. PMID: 16467788. Bibcode: 2006Natur.441..210S.
- ↑ Marta Barluenga, Kai N. Stölting, Walter Salzburger, Moritz Muschick i inni. Sympatric speciation in Nicaraguan crater lake cichlid fish. „Nature”. 439 (7077), s. 719–723, 2006-02-09. London: Nature Publishing Group. DOI: 10.1038/nature04325. ISSN 0028-0836. PMID: 16467837. Bibcode: 2006Natur.439..719B.
- ↑ Sergey Gavrilets. The Maynard Smith model of sympatric speciation. „Journal of Theoretical Biology”. 239 (2), s. 172–182, 2006-03-21. Amsterdam, Holandia: Elsevier. DOI: 10.1016/j.jtbi.2005.08.041. ISSN 0022-5193. PMID: 16242727.
- ↑ Troy E. Wood, Naoki Takebayashi, Michael S. Barker, Itay Mayrose i inni. The frequency of polyploid speciation in vascular plants. „Proc. Natl. Acad. Sci. U.S.A.”. 106 (33), s. 13875–13879, 18 sierpnia 2009. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0811575106. ISSN 0027-8424. PMID: 19667210. PMCID: PMC2728988. Bibcode: 2009PNAS..10613875W.
- ↑ Matthew J. Hegarty, Simon J. Hiscock. Genomic Clues to the Evolutionary Success of Polyploid Plants. „Current Biology”. 18 (10), s. R435–R444, 20 maja 2008. Cambridge, MA: Cell Press. DOI: 10.1016/j.cub.2008.03.043. ISSN 0960-9822. PMID: 18492478.
- ↑ Mattias Jakobsson, Jenny Hagenblad, Simon Tavaré, Torbjörn Säll i inni. A Unique Recent Origin of the Allotetraploid Species Arabidopsis suecica: Evidence from Nuclear DNA Markers. „Molecular Biology and Evolution”. 23 (6), s. 1217–1231, 2006. Oxford: Oxford University Press on behalf of the Society for Molecular Biology and Evolution. DOI: 10.1093/molbev/msk006. ISSN 0737-4038. PMID: 16549398.
- ↑ Torbjörn Säll, Mattias Jakobsson, Christina Lind-Halldén, Christer Halldén. Chloroplast DNA indicates a single origin of the allotetraploid Arabidopsis suecica. „Journal of Evolutionary Biology”. 16 (5), s. 1019–1029, 2003. Hoboken, NJ: Wiley-Blackwell on behalf of the European Society for Evolutionary Biology. DOI: 10.1046/j.1420-9101.2003.00554.x. ISSN 1010-061X. PMID: 14635917.
- ↑ Kirsten Bomblies, Detlef Weigel. Arabidopsis – a model genus for speciation. „Current Opinion in Genetics & Development”. 17 (6), s. 500–504, 2007. Amsterdam, Holandia: Elsevier. DOI: 10.1016/j.gde.2007.09.006. ISSN 0959-437X. PMID: 18006296.
- ↑ Marie Sémon, Kenneth H. Wolfe. Consequences of genome duplication. „Current Opinion in Genetics & Development”. 17 (6), s. 505–512, 2007. Amsterdam, the Netherlands: Elsevier. DOI: 10.1016/j.gde.2007.09.007. ISSN 0959-437X. PMID: 18006297.
- ↑ Eldredge i Gould 1972 ↓, s. 82–115.
- ↑ Michael J. Benton. Diversification and extinction in the history of life. „Science”. 268 (5207), s. 52–58, 7 kwietnia 1995. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.7701342. ISSN 0036-8075. PMID: 7701342. Bibcode: 1995Sci...268...52B.
- ↑ David M. Raup. Biological extinction in Earth history. „Science”. 231 (4745), s. 1528–1533, 1986-03-28. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.11542058. ISSN 0036-8075. PMID: 11542058. Bibcode: 1986Sci...231.1528R.
- ↑ John C. Avise, Stephen P. Hubbell, Francisco J. Ayala. In the light of evolution II: Biodiversity and extinction. „Proc. Natl. Acad. Sci. U.S.A.”. 105 (Suppl. 1), s. 11453–11457, 2008-08-12. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0802504105. ISSN 0027-8424. PMID: 18695213. PMCID: PMC2556414. Bibcode: 2008PNAS..10511453A.
- ↑ a b c David M. Raup. The role of extinction in evolution. „Proc. Natl. Acad. Sci. U.S.A.”. 91 (15), s. 6758–6763, 19 lipca 1994. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.91.15.6758. ISSN 0027-8424. PMID: 8041694. PMCID: PMC44280. Bibcode: 1994PNAS...91.6758R.
- ↑ Michael J. Novacek, Elsa E. Cleland. The current biodiversity extinction event: scenarios for mitigation and recovery. „Proc. Natl. Acad. Sci. U.S.A.”. 98 (10), s. 5466–5470, 2001-08-08. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.091093698. ISSN 0027-8424. PMID: 11344295. PMCID: PMC33235. Bibcode: 2001PNAS...98.5466N.
- ↑ Stuart Pimm, Peter Raven, Alan Peterson, Çağan H. Şekercioğlu i inni. Human impacts on the rates of recent, present and future bird extinctions. „Proc. Natl. Acad. Sci. U.S.A.”. 103 (29), s. 10941–10946, 2006-07-18. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0604181103. ISSN 0027-8424. PMID: 16829570. PMCID: PMC1544153. Bibcode: 2006PNAS..10310941P.
- ↑ Anthony D. Barnosky, Paul L. Koch, Robert S. Feranec, Scott L. Wing i inni. Assessing the Causes of Late Pleistocene Extinctions on the Continents. „Science”. 306 (5693), s. 70–75, 1 października 2004. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.1101476. ISSN 0036-8075. PMID: 15459379. Bibcode: 2004Sci...306...70B.
- ↑ Owen T. Lewis. Climate change, species–area curves and the extinction crisis. „Philosophical Transactions of the Royal Society B: Biological Sciences”. 361 (1465), s. 163–171, 2006-01-29. London: Royal Society. DOI: 10.1098/rstb.2005.1712. ISSN 0962-8436. PMID: 16553315. PMCID: PMC1831839.
- ↑ David Jablonski. Lessons from the past: Evolutionary impacts of mass extinctions. „Proc. Natl. Acad. Sci. U.S.A.”. 98 (10), s. 5393–5398, 2001-08-08. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.101092598. ISSN 0027-8424. PMID: 11344284. PMCID: PMC33224. Bibcode: 2001PNAS...98.5393J.
- ↑ Laurence A. Moran: Evolution and Abiogenesis. University of Toronto, 2006. [dostęp 2016-08-11]. [zarchiwizowane z tego adresu]. (ang.).
- ↑ Austin Cline: Abiogenesis & Evolution. atheism.about.com. [dostęp 2016-08-11]. (ang.).
- ↑ Evolution vs. Abiogenesis – Know the Difference!. Thinking Through Christianity, 2011-01-06. [dostęp 2016-08-11]. (ang.).
- ↑ Harmke Kamminga. Historical perspective: The problem of the origin of life in the context of developments in biology. „Origins of life and evolution of the biosphere”. 18 (1), s. 1–11, marzec 1988. Kluwer Academic Publishers. DOI: 10.1007/BF01808777. ISSN 1573-0875.
- ↑ Age of the Earth, United States Geological Survey, 9 lipca 2007 [dostęp 2015-05-31].
- ↑ Dalrymple 2001 ↓, s. 205–221.
- ↑ Gérard Manhesa, Claude J. Allègre, Bernard Dupréa, Bruno Hamelin. Lead isotope study of basic-ultrabasic layered complexes: Speculations about the age of the earth and primitive mantle characteristics. „Earth and Planetary Science Letters”. 47 (3), s. 370–382, 1980. Amsterdam, Holandia: Elsevier. DOI: 10.1016/0012-821X(80)90024-2. ISSN 0012-821X. Bibcode: 1980E&PSL..47..370M.
- ↑ Raven i Johnson 2002 ↓, s. 68.
- ↑ McKinney 1997 ↓, s. 110.
- ↑ Stearns i Stearns 1999 ↓, s. PA1921 x.
- ↑ Juli Peretó. Controversies on the origin of life. „International Microbiology”. 8 (1), s. 23–31, 2005. Barcelona: Spanish Society for Microbiology. ISSN 1139-6709. PMID: 15906258.
- ↑ Gerald F. Joyce. The antiquity of RNA-based evolution. „Nature”. 418 (6894), s. 214–221, 2002-07-11. London: Nature Publishing Group. DOI: 10.1038/418214a. ISSN 0028-0836. PMID: 12110897. Bibcode: 2002Natur.418..214J.
- ↑ Jack T. Trevors, Roland Psenner. From self-assembly of life to present-day bacteria: a possible role for nanocells. „FEMS Microbiology Reviews”. 25 (5), s. 573–582, 2001. Amsterdam, the Netherlands: Elsevier on behalf of the Federation of European Microbiological Societies. DOI: 10.1111/j.1574-6976.2001.tb00592.x. ISSN 1574-6976. PMID: 11742692.
- ↑ Douglas L. Theobald. A formal test of the theory of universal common ancestry. „Nature”. 465 (7295), s. 219–222, 2010-05-13. London: Nature Publishing Group. DOI: 10.1038/nature09014. ISSN 0028-0836. PMID: 20463738. Bibcode: 2010Natur.465..219T.
- ↑ Eric Bapteste, David A. Walsh. Does the ‘Ring of Life’ ring true?. „Trends in Microbiology”. 13 (6), s. 256–261, 2005. Cambridge, MA: Cell Press. DOI: 10.1016/j.tim.2005.03.012. ISSN 0966-842X. PMID: 15936656.
- ↑ Darwin 1859 ↓, s. 1.
- ↑ W. Ford Doolittle, Eric Bapteste. Pattern pluralism and the Tree of Life hypothesis. „Proc. Natl. Acad. Sci. U.S.A.”. 104 (7), s. 2043–2049, 2007-02-13. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.0610699104. ISSN 0027-8424. PMID: 17261804. PMCID: PMC1892968. Bibcode: 2007PNAS..104.2043D.
- ↑ Victor Kunin, Leon Goldovsky, Nikos Darzentas, Christos A. Ouzounis. The net of life: Reconstructing the microbial phylogenetic network. „Genome Research”. 15 (7), s. 954–959, 2005. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. DOI: 10.1101/gr.3666505. ISSN 1088-9051. PMID: 15965028. PMCID: PMC1172039.
- ↑ David Jablonski. The Future of the Fossil Record. „Science”. 284 (5423), s. 2114–2116, 1999-06-25. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.284.5423.2114. ISSN 0036-8075. PMID: 10381868.
- ↑ Stephen F. Mason. Origins of biomolecular handedness. „Nature”. 311 (5981), s. 19–23, 1984-09-06. London: Nature Publishing Group. DOI: 10.1038/311019a0. ISSN 0028-0836. PMID: 6472461. Bibcode: 1984Natur.311...19M.
- ↑ Yuri I. Wolf, Igor B. Rogozin, Nick V. Grishin, Eugene V. Koonin. Genome trees and the tree of life. „Trends in Genetics”. 18 (9), s. 472–479, 2002-09-01. Cambridge, MA: Cell Press. DOI: 10.1016/S0168-9525(02)02744-0. ISSN 0168-9525. PMID: 12175808.
- ↑ Ajit Varki, Tasha K. Altheide. Comparing the human and chimpanzee genomes: searching for needles in a haystack. „Genome Research”. 15 (12), s. 1746–1758, 2005. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. DOI: 10.1101/gr.3737405. ISSN 1088-9051. PMID: 16339373.
- ↑ Asger Hobolth, Ole F Christensen, Thomas Mailund & Mikkel H Schierup. Genomic Relationships and Speciation Times of Human, Chimpanzee, and Gorilla Inferred from a Coalescent Hidden Markov Model. „PloS Genetics”. 3, s. e7, 2007. DOI: 10.1371/journal.pgen.0030007. (ang.).
- ↑ Francesca D. Ciccarelli, Tobias Doerks, Christian von Mering, Christopher J. Creevey i inni. Toward Automatic Reconstruction of a Highly Resolved Tree of Life. „Science”. 311 (5765), s. 1283–1287, 2006-03-03. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.1123061. ISSN 0036-8075. PMID: 16513982. Bibcode: 2006Sci...311.1283C.
- ↑ a b Thomas Cavalier-Smith. Cell evolution and Earth history: stasis and revolution. „Philosophical Transactions of the Royal Society B: Biological Sciences”. 361 (1470), s. 969–1006, 2006-06-29. London: Royal Society. DOI: 10.1098/rstb.2006.1842. ISSN 0962-8436. PMID: 16754610. PMCID: PMC1578732.
- ↑ J. William Schopf. Fossil evidence of Archaean life. „Philosophical Transactions of the Royal Society B: Biological Sciences”. 361 (1470), s. 869–885, 2006-06-29. London: Royal Society. DOI: 10.1098/rstb.2006.1834. ISSN 0962-8436. PMID: 16754604. PMCID: PMC1578735.
- ↑ Władysław Altermann, Józef Kaźmierczak. Archean microfossils: a reappraisal of early life on Earth. „Research in Microbiology”. 154 (9), s. 611–617, 2003. Amsterdam, the Netherlands: Elsevier for the Pasteur Institute. DOI: 10.1016/j.resmic.2003.08.006. ISSN 0923-2508. PMID: 14596897.
- ↑ J. William Schopf. Disparate rates, differing fates: tempo and mode of evolution changed from the Precambrian to the Phanerozoic. „Proc. Natl. Acad. Sci. U.S.A.”. 91 (15), s. 6735–6742, 1994-07-19. Washington, D.C.: National Academy of Sciences. DOI: 10.1073/pnas.91.15.6735. ISSN 0027-8424. PMID: 8041691. PMCID: PMC44277. Bibcode: 1994PNAS...91.6735S.
- ↑ Anthony M. Poole, David Penny. Evaluating hypotheses for the origin of eukaryotes. „BioEssays”. 29 (1), s. 74–84, 2007. Hoboken, NJ: John Wiley & Sons. DOI: 10.1002/bies.20516. ISSN 0265-9247. PMID: 17187354.
- ↑ Sabrina D. Dyall, Mark T. Brown, Patricia J. Johnson. Ancient Invasions: From Endosymbionts to Organelles. „Science”. 304 (5668), s. 253–257, 2004-04-09. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.1094884. ISSN 0036-8075. PMID: 15073369. Bibcode: 2004Sci...304..253D.
- ↑ William Martin. The missing link between hydrogenosomes and mitochondria. „Trends in Microbiology”. 13 (10), s. 457–459, 2005. Cambridge, MA: Cell Press. DOI: 10.1016/j.tim.2005.08.005. ISSN 0966-842X. PMID: 16109488.
- ↑ B. Lang, Michael W. Gray, Gertraud Burger. Mitochondrial genome evolution and the origin of eukaryotes. „Annual Review of Genetics”. 33, s. 351–397, 1999. Palo Alto, CA: Annual Reviews. DOI: 10.1146/annurev.genet.33.1.351. ISSN 0066-4197. PMID: 10690412.
- ↑ Geoffrey Ian McFadden. Endosymbiosis and evolution of the plant cell. „Current Opinion in Plant Biology”. 2 (6), s. 513–519, 1999-12-01. Amsterdam, Holandia: Elsevier. DOI: 10.1016/S1369-5266(99)00025-4. ISSN 1369-5266. PMID: 10607659.
- ↑ Edward F. DeLong, Norman R. Pace. Environmental Diversity of Bacteria and Archaea. „Systematic Biology”. 50 (4), s. 470–478, 1 sierpnia 2001. Oxford: Oxford University Press on behalf of the Society of Systematic Biologists. DOI: 10.1080/106351501750435040. ISSN 1063-5157. PMID: 12116647.
- ↑ Dale Kaiser. Building a multicellular organism. „Annual Review of Genetics”. 35, s. 103–123, grudzień 2001. Palo Alto, CA: Annual Reviews. DOI: 10.1146/annurev.genet.35.102401.090145. ISSN 0066-4197. PMID: 11700279.
- ↑ Carl Zimmer: Genetic Flip Helped Organisms Go From One Cell to Many. [w:] The New York Times [on-line]. 2016-01-07. [dostęp 2016-01-07].
- ↑ James W. Valentine, David Jablonski, Douglas H. Erwin. Fossils, molecules and embryos: new perspectives on the Cambrian explosion. „Development”. 126 (5), s. 851–859, 1999-03-01. Cambridge: The Company of Biologists. ISSN 0950-1991. PMID: 9927587. [dostęp 2014-12-30].
- ↑ Susumu Ohno. The reason for as well as the consequence of the Cambrian explosion in animal evolution. „Journal of Molecular Evolution”. 44 (Suppl. 1), s. S23–S27, 1997. New York: Springer-Verlag New York. DOI: 10.1007/PL00000055. ISSN 0022-2844. PMID: 9071008.
- ↑ James W. Valentine, David Jablonski. Morphological and developmental macroevolution: a paleontological perspective. „The International Journal of Developmental Biology”. 47 (7–8), s. 517–522, 2003. Bilbao, Spain: University of the Basque Country Press. ISSN 0214-6282. PMID: 14756327. [dostęp 2014-12-30].
- ↑ Elizabeth R. Waters. Molecular adaptation and the origin of land plants. „Molecular Phylogenetics and Evolution”. 29 (3), s. 456–463, 2003. Academic Press. DOI: 10.1016/j.ympev.2003.07.018. ISSN 1055-7903. PMID: 14615186.
- ↑ Peter J. Mayhew. Why are there so many insect species? Perspectives from fossils and phylogenies. „Biological Reviews”. 82 (3), s. 425–454, 2007. Cambridge: Cambridge University Press on behalf of the Cambridge Philosophical Society. DOI: 10.1111/j.1469-185X.2007.00018.x. ISSN 1464-7931. PMID: 17624962.
- ↑ Robert L. Carroll. The Palaeozoic Ancestry of Salamanders, Frogs and Caecilians. „Zoological Journal of the Linnean Society”. 150 (Supplement s1), s. 1–140, 2007. Hoboken, NJ: Wiley-Blackwell. DOI: 10.1111/j.1096-3642.2007.00246.x. ISSN 1096-3642. PMID: 12752770.
- ↑ John R. Wible, Guillermo W. Rougier, Michael J. Novacek, Robert J. Asher. Cretaceous eutherians and Laurasian origin for placental mammals near the K/T boundary. „Nature”. 447 (7147), s. 1003–1006, 2007-06-21. London: Nature Publishing Group. DOI: 10.1038/nature05854. ISSN 0028-0836. PMID: 17581585. Bibcode: 2007Natur.447.1003W.
- ↑ Lawrence M. Witmer. Palaeontology: An icon knocked from its perch. „Nature”. 475 (7357), s. 458–459, 28 lipca 2011. London: Nature Publishing Group. DOI: 10.1038/475458a. ISSN 0028-0836. PMID: 21796198.
- ↑ James J. Bull, Holly A. Wichman. Applied evolution. „Annual Review of Ecology and Systematics”. 32, s. 183–217, 2001. Palo Alto, CA: Annual Reviews. DOI: 10.1146/annurev.ecolsys.32.081501.114020. ISSN 1545-2069.
- ↑ John F. Doebley, Brandon S. Gaut, Bruce D. Smith. The Molecular Genetics of Crop Domestication. „Cell”. 127 (7), s. 1309–1321, 2006-12-29. Cambridge, MA: Cell Press. DOI: 10.1016/j.cell.2006.12.006. ISSN 0092-8674. PMID: 17190597.
- ↑ Christian Jäckel, Peter Kast, Donald Hilvert. Protein Design by Directed Evolution. „Annual Review of Biophysics”. 37, s. 153–173, 2008. Palo Alto, CA: Annual Reviews. DOI: 10.1146/annurev.biophys.37.032807.125832. ISSN 1936-122X. PMID: 18573077.
- ↑ Brendan Maher. Evolution: Biology’s next top model?. „Nature”. 458 (7239), s. 695–698, 8 kwietnia 2009. London: Nature Publishing Group. DOI: 10.1038/458695a. ISSN 0028-0836. PMID: 19360058.
- ↑ Richard Borowsky. Restoring sight in blind cavefish. „Current Biology”. 18 (1), s. R23–R24, 2008-01-08. Cambridge, MA: Cell Press. DOI: 10.1016/j.cub.2007.11.023. ISSN 0960-9822. PMID: 18177707.
- ↑ Joshua B. Gross, Richard Borowsky, Clifford J. Tabin, Gregory S. Barsh. A novel role for Mc1r in the parallel evolution of depigmentation in independent populations of the cavefish Astyanax mexicanus. „PLOS Genetics”. 5 (1), s. e1000326, 2009-01-02. San Francisco, CA: Public Library of Science. DOI: 10.1371/journal.pgen.1000326. ISSN 1553-7390. PMID: 19119422. PMCID: PMC2603666.
- ↑ Lauren M.F. Merlo, John W. Pepper, Brian J. Reid, Carlo C. Maley. Cancer as an evolutionary and ecological process.. „Nature Reviews Cancer”. 6 (12), s. 924–935, 2006. London: Nature Publishing Group. DOI: 10.1038/nrc2013. ISSN 1474-175X. PMID: 17109012.
- ↑ Dabo Pan, Weiwei Xue, Wenqi Zhang, Huanxiang Liu i inni. Understanding the drug resistance mechanism of hepatitis C virus NS3/4A to ITMN-191 due to R155K, A156V, D168A/E mutations: a computational study.. „Biochimica et Biophysica Acta (BBA) – General Subjects”. 1820 (10), s. 1526–1534, 2012. Amsterdam, Holandia: Elsevier. DOI: 10.1016/j.bbagen.2012.06.001. ISSN 0304-4165. PMID: 22698669.
- ↑ Neil Woodford, Matthew J. Ellington. The emergence of antibiotic resistance by mutation. „Clinical Microbiology and Infection”. 13 (1), s. 5–18, 2007. Amsterdam, Holandia: Elsevier for the European Society of Clinical Microbiology and Infectious Diseases. DOI: 10.1111/j.1469-0691.2006.01492.x. ISSN 1198-743X. PMID: 17184282.
- ↑ Pierrick Labbé, Claire Berticat, Arnaud Berthomieu, Sandra Unal i inni. Forty Years of Erratic Insecticide Resistance Evolution in the Mosquito Culex pipiens. „PLOS Genetics”. 3 (11), s. e205, 2007-11-16. San Francisco, CA: Public Library of Science. DOI: 10.1371/journal.pgen.0030205. ISSN 1553-7390. PMID: 18020711.
- ↑ Paul Neve. Challenges for herbicide resistance evolution and management: 50 years after Harper. „Weed Research”. 47 (5), s. 365–369, październik 2007. Hoboken, NJ: Wiley-Blackwell on behalf of the European Weed Research Society. DOI: 10.1111/j.1365-3180.2007.00581.x. ISSN 0043-1737.
- ↑ Alexandro Rodríguez-Rojas, Jerónimo Rodríguez-Beltrán, Alejandro Couce, Jesús Blázquez. Antibiotics and antibiotic resistance: A bitter fight against evolution. „International Journal of Medical Microbiology”. 303 (6–7), s. 293–297, sierpień 2013. Amsterdam, Holandia: Elsevier. DOI: 10.1016/j.ijmm.2013.02.004. ISSN 1438-4221. PMID: 23517688.
- ↑ Martijn F. Schenk, Ivan G. Szendro, Joachim Krug, J. Arjan G. M. de Visser. Quantifying the Adaptive Potential of an Antibiotic Resistance Enzyme. „PLOS Genetics”. 8 (6), s. e1002783, 2012-06-28. San Francisco, CA: Public Library of Science. DOI: 10.1371/journal.pgen.1002783. ISSN 1553-7390. PMID: 22761587.
- ↑ Andrew F. Read, Penelope A. Lynch, Matthew B. Thomas. How to Make Evolution-Proof Insecticides for Malaria Control. „PLOS Biology”. 7 (4), s. e1000058, 2009-04-07. San Francisco, CA: Public Library of Science. DOI: 10.1371/journal.pbio.1000058. ISSN 1545-7885. PMID: 19355786.
- ↑ Alex S. Fraser. Monte Carlo Analyses of Genetic Models. „Nature”. 181 (4603), s. 208–209, 1958-01-18. London: Nature Publishing Group. DOI: 10.1038/181208a0. ISSN 0028-0836. PMID: 13504138. Bibcode: 1958Natur.181..208F.
- ↑ Rechenberg 1973 ↓.
- ↑ Holland 1975 ↓.
- ↑ Koza 1992 ↓.
- ↑ Mo Jamshidi. Tools for intelligent control: fuzzy controllers, neural networks and genetic algorithms. „Philosophical Transactions of the Royal Society A”. 361 (1809), s. 1781–1808, 2003-08-15. London: Royal Society. DOI: 10.1098/rsta.2003.1225. ISSN 1364-503X. PMID: 12952685. Bibcode: 2003RSPTA.361.1781J.
- ↑ Browne 2003 ↓, s. 376–379.
- ↑ Chaberek 2012 ↓, s. 82.
- ↑ St. G. Mivart: On the Genesis of Species. Nowy Jork: D. Appleton and Company, 1871, s. 74.
- ↑ a b Bowler 2003 ↓.
- ↑ W wydaniu z 1859 r. na s. 488.
- ↑ Human evolution: A brief history of the research since Darwin’s time. W: Biological Evolution: Facts and Theories. A Critical Appraisal 150 Years After „The Origin of Species” (International Conference at Gregorian University). G. Auletta, M. Leclerc, R.A. Martínez (red.). Rzym: Gregorian and Biblical Press, 2009. ISBN 978-88-7839-180-2.
- ↑ Por. Theories of Evolution. W: A Century of Darwin. S.A. Barnett i inni (red.). Londyn: 1962, s. 10–18.
- ↑ J. Huxley: The Evolutionary Vision: The Convocation Address. W: Evolution after Darwin. The University of Chicago Centennial. Sol Tax i in. (red.). Chicago: The University of Chicago Press, 1960, s. 249-216.
- ↑ Chaberek 2012 ↓, s. 67.
- ↑ Warszawa:PIW, 1997.
- ↑ Warszawa 2007.
- ↑ „Kościół wobec ewolucji”, 26 listopada 2006, radiomaryja.pl. radiomaryja.pl. [zarchiwizowane z tego adresu (2009-02-15)].
- ↑ Por. M. Chaberek: Drugi okres (1950-2011) – umiarkowana akceptacja teistycznego ewolucjonizmu. W: Tenże: Kościół a ewolucja. Warszawa: Fronda, 2012, s. 221–347. ISBN 978-83-62268-30-6.
- ↑ Dennett 1995 ↓.
- ↑ Section Three: The Origins of Evolutionary Theory. W: Ian C. Johnston: ... And Still We Evolve: A Handbook for the Early History of Modern Science. Wyd. 3. Nanaimo, BC: Liberal Studies Department, Malaspina University-College, 1999. [dostęp 2015-01-01].
- ↑ Emile Zuckerkandl. Intelligent design and biological complexity. „Gene”. 385, s. 2–18, 2006-12-30. Amsterdam, the Netherlands: Elsevier. DOI: 10.1016/j.gene.2006.03.025. ISSN 0378-1119. PMID: 17011142.
- ↑ Chaberek 2012 ↓, s. 46–51.
- ↑ Jon D. Miller, Eugenie C. Scott, Shinji Okamoto. Public Acceptance of Evolution. „Science”. 313 (5788), s. 765–766, 2006-08-11. Washington, D.C.: American Association for the Advancement of Science. DOI: 10.1126/science.1126746. ISSN 0036-8075. PMID: 16902112.
- ↑ David Nathaniel Spergel, Licia Verde, Hiranya V. Peiris, Eiichiro Komatsu i inni. First-Year Wilkinson Microwave Anisotropy Probe (WMAP) Observations: Determination of Cosmological Parameters. „The Astrophysical Journal Supplement Series”. 148 (1), s. 175–194, 2003. Chicago, IL: University of Chicago Press on behalf of the American Astronomical Society. DOI: 10.1086/377226. arXiv:astro-ph/0302209. Bibcode: 2003ApJS..148..175S.
- ↑ Simon A. Wilde, John W. Valley, William H. Peck, Colin M. Graham. Evidence from detrital zircons for the existence of continental crust and oceans on the Earth 4.4 Gyr ago. „Nature”. 409 (6817), s. 175–178, 11 stycznia 2001. London: Nature Publishing Group. DOI: 10.1038/35051550. ISSN 0028-0836. PMID: 11196637.
- ↑ Steve Stewart-Williams: Darwin, Bóg i Sens Życia. Stare Groszki: Wydawnictwo CiS, 2014, s. 58. ISBN 978-83-61710-12-7.
- ↑ a b Glenn Branch. Understanding Creationism after Kitzmiller. „BioScience”. 57 (3), s. 278–284, 2007. Oxford: Oxford University Press on behalf of the American Institute of Biological Sciences. DOI: 10.1641/B570313. ISSN 0006-3568.
- ↑ Michael J. Dougherty. Is the human race evolving or devolving?. „Scientific American”, 10 lipca 1998. Stuttgart: Georg von Holtzbrinck Publishing Group. ISSN 0036-8733. [dostęp 2015-09-11].
- ↑ Lane 1996 ↓, s. 61.
- ↑ Józef Życiński: Cel i konieczność w ewolucji. W: Michał Heller, Józef Życiński: Dylematy ewolucji. Kraków: Copernicus Center Press, 2016, s. 117–118, seria: Nauka i Religia. ISBN 978-83-7886-212-3.
- ↑ Futuyma 2008 ↓, s. 9.
- ↑ a b Finding Design in Nature. [w:] New York Times [on-line]. 7 lipca 2005. [dostęp 2017-02-14].
- ↑ Chaberek 2012 ↓, s. 320–321.
- ↑ Schönborn 2009 ↓.
- ↑ Michał Heller: Filozofia przypadku. Kosmiczna fuga z preludium i codą. Kraków: Copernicus Center, 2015. ISBN 978-83-7886-158-4.
- ↑ Kent Hovind, 2nd Kaw of Thermodynamics [online].
- ↑ Termodynamika podważa ewolucję [online], PCh24.pl.
- ↑ John Rennie, 15 Answers to Creationist Nonsense [online], scientificamerican, 1 lipca 2002.
Bibliografia
- Wolfgang Banzhaf, Frank H. Eeckman, Genome growth and the evolution of the genotype-phenotype map, „Evolution and Biocomputation; Lecture Notes in Computer Science”, 899, Berlin; New York: Springer-Verlag Berlin Heidelberg, 1995, DOI: 10.1007/3-540-59046-3_11, ISBN 3-540-59046-3, LCCN 95005970, ISSN 0302-9743, OCLC 32049812.
- Francisco J. Ayala, John C. Avise: Essential Readings in Evolutionary Biology. Baltimore, MD: Johns Hopkins University Press, 2014. ISBN 978-1-4214-1305-1. LCCN 2013027718. OCLC 854285705.
- John A. Birdsell, Christopher Wills, The Evolutionary Origin and Maintenance of Sexual Recombination: A Review of Contemporary Models, [w:] Ross J. MacIntyre, Michael T. Clegg, Evolutionary Biology, New York: Springer Science+Business Media, 2003, ISBN 978-1-4419-3385-0, ISSN 0071-3260, OCLC 751583918.
- Peter J. Bowler: The Mendelian Revolution: The Emergence of Hereditarian Concepts in Modern Science and Society. Baltimore, MD: Johns Hopkins University Press, 1989. ISBN 0-8018-3888-6. LCCN 89030914. OCLC 19322402.
- Peter J. Bowler: Evolution: The History of an Idea. Wyd. 3rd completely rev. and expanded. Berkeley, CA: University of California Press, 2003. ISBN 0-520-23693-9. LCCN 2002007569. OCLC 49824702.
- Janet Browne: Charles Darwin: The Power of Place wolumin 2. London: Pimlico, 2003. ISBN 0-7126-6837-3. LCCN 94006598. OCLC 52327000.
- Frederick Burkhardt, Sydney Smith: The Correspondence of Charles Darwin. T. 7. Cambridge: Cambridge University Press, 1991, s. 1858–1859, seria: The Correspondence of Charles Darwin. ISBN 0-521-38564-4. LCCN 84045347. OCLC 185662993.
- Sean B. Carroll, Jennifer K. Grenier, Scott D. Weatherbee: From DNA to Diversity: Molecular Genetics and the Evolution of Animal Design. Wyd. 2nd. Malden, MA: Blackwell Publishing, 2005. ISBN 1-4051-1950-0. LCCN 2003027991. OCLC 53972564.
- Michał Chaberek: Kościół a ewolucja. Warszawa: Fronda, 2012, s. 382. ISBN 978-83-62268-30-6.
- Jerry A. Coyne: Why Evolution is True. New York: Viking, 2009. ISBN 978-0-670-02053-9. LCCN 2008033973. OCLC 233549529.
- Joel Cracraft, Rodger W. Bybee: Evolutionary Science and Society: Educating a New Generation. Colorado Springs, CO: Biological Sciences Curriculum Study, 2005. ISBN 1-929614-23-3. OCLC 64228003. [dostęp 2014-12-06]. „Revised Proceedings of the BSCS, AIBS Symposium November 2004, Chicago, IL”
- G. Brent Dalrymple, The age of the Earth in the twentieth century: a problem (mostly) solved, [w:] C.L.E. Lewis, S.J. Knell, The Age of the Earth: from 4004 BC to AD 2002, „Geological Society Special Publication wolumin 190”, London: Geological Society of London, 2001, DOI: 10.1144/gsl.sp.2001.190.01.14, ISBN 1-86239-093-2, LCCN 2003464816, ISSN 0305-8719, Bibcode: 2001GSLSP.190..205D, OCLC 48570033.
- Charles Darwin: On the Origin of Species by Means of Natural Selection, or the Preservation of Favoured Races in the Struggle for Life. Wyd. 1st. London: John Murray, 1859. LCCN 06017473. OCLC 741260650. The book is available from The Complete Work of Charles Darwin Online. Retrieved 2014-11-21.
- Charles Darwin: The Expression of the Emotions in Man and Animals. London: John Murray, 1872. LCCN 04002793. OCLC 1102785.
- Francis Darwin: The foundations of The origin of species, a sketch written in 1842. Cambridge: Printed at the University Press, 1909. LCCN 61057537. OCLC 1184581. [dostęp 2014-11-27].
- Richard Dawkins: The Blind Watchmaker. London: Penguin Books, 1990, seria: Penguin Science. ISBN 0-14-014481-1. OCLC 60143870.
- Daniel Dennett: Darwin’s Dangerous Idea: Evolution and the Meanings of Life. New York: Simon & Schuster, 1995. ISBN 0-684-80290-2. LCCN 94049158. OCLC 31867409.
- Theodosius Dobzhansky: On Some Fundamental Concepts of Darwinian Biology. W: Theodosius Dobzhansky, Max K. Hecht, William C. Steere: Evolutionary Biology. Volume 2. Wyd. 1st. New York: Appleton-Century-Crofts, 1968. DOI: 10.1007/978-1-4684-8094-8_1. OCLC 24875357.
- Theodosius Dobzhansky: Genetics of the Evolutionary Process. New York: Columbia University Press, 1970. ISBN 0-231-02837-7. LCCN 72127363. OCLC 97663.
- Niles Eldredge, Stephen Jay Gould: Punctuated equilibria: an alternative to phyletic gradualism. W: Thomas J.M. Schopf: Models in Paleobiology. San Francisco, CA: Freeman, Cooper, 1972. ISBN 0-87735-325-5. LCCN 72078387. OCLC 572084.
- Niles Eldredge: Time Frames: The Rethinking of Darwinian Evolution and the Theory of Punctuated Equilibria. New York: Simon & Schuster, 1985. ISBN 0-671-49555-0. LCCN 84023632. OCLC 11443805.
- Warren J. Ewens: Mathematical Population Genetics. Wyd. 2nd. T. I: Theoretical Introduction. New York: Springer-Verlag New York, 2004, seria: Interdisciplinary Applied Mathematics. ISBN 0-387-20191-2. LCCN 2003065728. OCLC 53231891.
- Douglas J. Futuyma: The Fruit of the Tree of Life: Insights into Evolution and Ecology. W: Joel Cracraft, Michael J. Donoghue: Assembling the Tree of Life. Oxford; New York: Oxford University Press, 2004. ISBN 0-19-517234-5. LCCN 2003058012. OCLC 61342697. „Proceedings of a symposium held at the American Museum of Natural History in New York, 2002.”
- Douglas J. Futuyma: Evolution. Sunderland, MA: Sinauer Associates, 2005. ISBN 0-87893-187-2. LCCN 2004029808. OCLC 57311264.
- Douglas J. Futuyma: Ewolucja. Wydawnictwo Uniwersytetu Warszawskiego, 2008. ISBN 978-83-235-0577-8.
- Stephen Jay Gould: The Structure of Evolutionary Theory. Cambridge, MA: Belknap Press of Harvard University Press, 2002. ISBN 0-674-00613-5. LCCN 2001043556. OCLC 47869352.
- Peter Gray: Psychology. Wyd. 5th. New York: Worth Publishers, 2007. ISBN 978-0-7167-0617-5. LCCN 2006921149. OCLC 76872504.
- Brian K. Hall, Benedikt Hallgrímsson: Strickberger’s Evolution. Wyd. 4th. Sudbury, MA: Jones and Bartlett Publishers, 2008. ISBN 978-0-7637-0066-9. LCCN 2007008981. OCLC 85814089.
- Willi Hennig: Phylogenetic Systematics. Wyd. Reissue. Urbana, IL: University of Illinois Press, 1999. ISBN 0-252-06814-9. LCCN 78031969. OCLC 722701473.
- John H. Holland: Adaptation in Natural and Artificial Systems: An Introductory Analysis with Applications to Biology, Control, and Artificial Intelligence. Ann Arbor, MI: University of Michigan Press, 1975. ISBN 0-472-08460-7. LCCN 74078988. OCLC 1531617.
- Eva Jablonka, Marion J. Lamb: Evolution in Four Dimensions: Genetic, Epigenetic, Behavioral, and Symbolic Variation in the History of Life. Anna Zeligowski (ilustracje). Cambridge, MA: MIT Press, 2005. ISBN 0-262-10107-6. LCCN 2004058193. OCLC 61896061.
- Kostas Kampourakis: Understanding Evolution. Cambridge; New York: Cambridge University Press, 2014. ISBN 978-1-107-03491-4. LCCN 2013034917. OCLC 855585457.
- Geoffrey Kirk, John Raven, Malcolm Schofield: The Presocratic Philosophers: A Critical History with a Selection of Texts. Wyd. 2nd. Cambridge; New York: Cambridge University Press, 1983. ISBN 0-521-27455-9. LCCN 82023505. OCLC 9081712.
- John R. Koza: Genetic Programming: On the Programming of Computers by Means of Natural Selection. Cambridge, MA: MIT Press, 1992, seria: Complex Adaptive Systems. ISBN 0-262-11170-5. LCCN 92025785. OCLC 26263956.
- Jean-Baptiste Lamarck: Philosophie Zoologique. Paris: Dentu et L’Auteur, 1809. OCLC 2210044. Retrieved 2014-11-29.
- David H. Lane: The Phenomenon of Teilhard: Prophet for a New Age. Wyd. 1st. Macon, GA: Mercer University Press, 1996. ISBN 0-86554-498-0. LCCN 96008777. OCLC 34710780.
- Lois N. Magner: A History of the Life Sciences. Wyd. 3rd rev. and expanded. New York: Marcel Dekker, 2002. ISBN 0-8247-0824-5. LCCN 2002031313. OCLC 50410202.
- Stephen F. Mason: A History of the Sciences. Wyd. New rev. New York: Collier Books, 1962, seria: Collier Books. Science Library, CS9. LCCN 62003378. OCLC 568032626.
- John Maynard Smith: The Evolution of Sex. Cambridge; New York: Cambridge University Press, 1978. ISBN 0-521-29302-2. LCCN 77085689. OCLC 3413793.
- John Maynard Smith, The Units of Selection, [w:] Gregory R. Bock, Jamie A. Goode, The Limits of Reductionism in Biology, „Novartis Foundation Symposia”, 213, Chichester, England: John Wiley & Sons, 1998, s. 203–11, DOI: 10.1002/9780470515488.ch15, ISBN 0-471-97770-5, LCCN 98002779, ISSN 1935-4657, PMID: 9653725, OCLC 38311600. „Papers from the Symposium on the Limits of Reductionism in Biology, held at the Novartis Foundation, London, May 13–15, 1997.”
- Ernst Mayr: Systematics and the Origin of Species from the Viewpoint of a Zoologist. T. 13. New York: Columbia University Press, 1942, seria: Columbia Biological Series. LCCN 43001098. OCLC 766053.
- Ernst Mayr: The Growth of Biological Thought: Diversity, Evolution, and Inheritance. Cambridge, MA: Belknap Press, 1982. ISBN 0-674-36445-7. LCCN 81013204. OCLC 7875904.
- Ernst Mayr: What Evolution Is. London: Weidenfeld & Nicolson, 2002, seria: Science Masters. ISBN 0-297-60741-3. LCCN 2001036562. OCLC 248107061.
- Michael L. McKinney: How do rare species avoid extinction? A paleontological view. W: William E. Kunin, Kevin J. Gaston: The Biology of Rarity: Causes and consequences of rare–common differences. Wyd. 1st. London; New York: Chapman & Hall, 1997. ISBN 0-412-63380-9. LCCN 96071014. OCLC 36442106.
- G. Tyler Miller, Scott E. Spoolman: Environmental Science. Wyd. 14th. Belmont, CA: Brooks/Cole, 2012. ISBN 978-1-111-98893-7. LCCN 2011934330. OCLC 741539226. [dostęp 2014-12-27].
- Randy Moore, Mark Decker, Sehoya Cotner: Chronology of the Evolution-Creationism Controversy. Santa Barbara, CA: Greenwood Press/ABC-CLIO, 2010. ISBN 978-0-313-36287-3. LCCN 2009039784. OCLC 422757410.
- Paul Nardon, Anne-Marie Grenier: Serial Endosymbiosis Theory and Weevil Evolution: The Role of Symbiosis. W: Lynn Margulis, René Fester: Symbiosis as a Source of Evolutionary Innovation: Speciation and Morphogenesis. Cambridge, MA: MIT Press, 1991. ISBN 0-262-13269-9. LCCN 90020439. OCLC 22597587.
- National Academy of Sciences, Institute of Medicine: Science, Evolution, and Creationism. Washington, D.C.: National Academy Press, 2008. ISBN 978-0-309-10586-6. LCCN 2007015904. OCLC 123539346. [dostęp 2014-11-22].
- Eugene P. Odum: Fundamentals of Ecology. Wyd. 3rd. Philadelphia, PA: Saunders, 1971. ISBN 0-7216-6941-7. LCCN 76081826. OCLC 154846.
- Samir Okasha: Evolution and the Levels of Selection. Oxford; New York: Oxford University Press, 2006. ISBN 0-19-926797-9. LCCN 2006039679. OCLC 70985413.
- Joseph Panno: The Cell: Evolution of the First Organism. New York: Facts on File, 2005, seria: Facts on File science library. ISBN 0-8160-4946-7. LCCN 2003025841. OCLC 53901436.
- Joram Piatigorsky i inni, Recruitment of enzymes and stress proteins as lens crystallins, [w:] Bengt Jansson i inni, Toward a Molecular Basis of Alcohol Use and Abuse, t. 71, Basel; Boston: Birkhäuser Verlag, 1994 (Experientia), s. 241–50, DOI: 10.1007/978-3-0348-7330-7_24, ISBN 3-7643-2940-8, LCCN 94010167, ISSN 1023-294X, PMID: 8032155, OCLC 30030941.
- William B. Provine: The Origins of Theoretical Population Genetics. Wyd. 2nd. Chicago, IL: University of Chicago Press, 1971, seria: Chicago History of Science and Medicine. ISBN 0-226-68464-4. LCCN 2001027561. OCLC 46660910.
- William B. Provine: Progress in Evolution and Meaning in Life. W: Matthew H. Nitecki: Evolutionary Progress. Chicago, IL: University of Chicago Press, 1988. ISBN 0-226-58693-6. LCCN 88020835. OCLC 18380658. „This book is the result of the Spring Systematics Symposium held in May, 1987, at the Field Museum in Chicago”
- David Quammen: The Reluctant Mr. Darwin: An Intimate Portrait of Charles Darwin and the Making of His Theory of Evolution. Wyd. 1st. New York: Atlas Books/W. W. Norton & Company, 2006, seria: Great Discoveries. ISBN 978-0-393-05981-6. LCCN 2006009864. OCLC 65400177.
- Peter H. Raven, George B. Johnson: Biology. Wyd. 6. Boston, MA: McGraw-Hill, 2002. ISBN 0-07-112261-3. LCCN 2001030052. OCLC 45806501.
- John Ray: Historia Plantarum (History of Plants). T. I. Londini: Typis Mariæ Clark, 1686. LCCN agr11000774. OCLC 2126030.
- Ingo Rechenberg: Evolutionsstrategie; Optimierung technischer Systeme nach Prinzipien der biologischen Evolution. T. 15. Stuttgart-Bad Cannstatt: Frommann-Holzboog, 1973, seria: Problemata. ISBN 3-7728-0373-3. LCCN 74320689. OCLC 9020616. (niem.).
- Matt Ridley: The Red Queen: Sex and the Evolution of Human Nature. New York: Viking, 1993. ISBN 0-670-84357-1. OCLC 636657988.
- Christoph Schönborn: Cel czy przypadek? Dzieło stworzenia i ewolucja z punktu widzenia racjonalnej wiary. Kielce: Jedność, 2009, s. 175. ISBN 978-83-7442-587-2.
- Beverly Peterson Stearns, Stephen C. Stearns: Watching, from the Edge of Extinction. New Haven, CT: Yale University Press, 1999. ISBN 0-300-07606-1. LCCN 98034087. OCLC 47011675. [dostęp 2014-12-27].
- Anthony Stevens: Archetype: A Natural History of the Self. London: Routledge & Kegan Paul, 1982. ISBN 0-7100-0980-1. LCCN 84672250. OCLC 10458367.
- Mary Jane West-Eberhard: Developmental Plasticity and Evolution. Oxford; New York: Oxford University Press, 2003. ISBN 0-19-512235-6. LCCN 2001055164. OCLC 48398911.
- E.O. Wiley, Bruce S. Lieberman: Phylogenetics: Theory and Practice of Phylogenetic Systematics. Wyd. 2nd. Hoboken, NJ: Wiley-Blackwell, 2011. DOI: 10.1002/9781118017883. ISBN 978-0-470-90596-8. LCCN 2010044283. OCLC 741259265.
- Sewall Wright: Genetic and Biometric Foundations. T. 1. Chicago, IL: University of Chicago Press, 1984, seria: Evolution and the Genetics of Populations. ISBN 0-226-91038-5. LCCN 67025533. OCLC 246124737.
Linki zewnętrzne
 Karolina Głowacka i Piotr Bernatowicz, Wspólnota życia – ewolucja odsłania prawdę o pochodzeniu człowieka, kanał „Radio Naukowe” na YouTube, 15 sierpnia 2024 [dostęp 2024-08-15].
Karolina Głowacka i Piotr Bernatowicz, Wspólnota życia – ewolucja odsłania prawdę o pochodzeniu człowieka, kanał „Radio Naukowe” na YouTube, 15 sierpnia 2024 [dostęp 2024-08-15].
![]() Artykuły na Stanford Encyclopedia of Philosophy (ang.) [dostęp 2018-01-29]:
Artykuły na Stanford Encyclopedia of Philosophy (ang.) [dostęp 2018-01-29]:
- Roberta L. Millstein, Evolution, 25 sierpnia 2017.
- Phillip Sloan, The Concept of Evolution to 1872, 3 czerwca 2014.

