Telomerase
| Telomerase | ||
|---|---|---|
| ||
| Bezeichner | ||
| Externe IDs |
| |
| Enzymklassifikation | ||
| EC, Kategorie | 2.7.7.49, Nukleotidyltransferase | |
| Substrat | DNA | |
| Produkte | Telomere der DNA | |
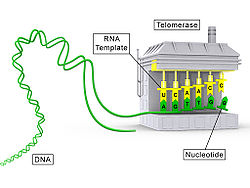
Die Telomerase ist ein Enzym des Zellkerns, welches aus einem Protein- (TERT) und einem langen RNA-Anteil (TR) besteht und somit ein Ribonukleoprotein ist. Dieses Enzym stellt die Endstücke der Chromosomen, die sogenannten Telomere, wieder her. Die Enzymaktivität der Telomerase lässt sich durch die TRAP-Methode feststellen.
Entdeckung
Die Telomerase wurde 1985 von den beiden Forscherinnen Elizabeth Blackburn und Carol Greider in dem Wimpertierchen Tetrahymena entdeckt.[1] Sie wurden dafür 2009 mit dem Paul-Ehrlich-Preis und, zusammen mit Jack W. Szostak, mit dem Nobelpreis für Physiologie und Medizin ausgezeichnet.
Merkmale
Das Enzym ist eine reverse Transkriptase (Telomerase-Reverse-Transkriptase, TERT), welche den RNA-Anteil (Telomerase-RNA, TR oder TERC) als Matrize (Vorlage) verwendet. Beim Menschen ist der RNA-Anteil (hTERC, mit h für human) beispielsweise die Sequenz 3'-CAAUCCCAAUC-5', was bedeutet, dass das Telomer jeweils um eine Sequenz d(5'-GGTTAG-3') verlängert wird (Präfix d meint DNA statt RNA).[2]
Funktion
Die Länge der Telomere ist bei verschiedenen Arten unterschiedlich. Bei Menschen beträgt die Länge etwa 10 kb (Kilobasen), nach mehreren Zellteilungen (etwa 50–100) kann eine Länge von ca. 4–6 kb erreicht werden. Danach geht die Zelle in die Ruhephase über und teilt sich nicht mehr. Bei jeder Zellteilung geht ein Stück (ca. 100 Nukleotide) der Telomere verloren. Die Telomerase verhindert in bestimmten Zellen durch die Wiederherstellung der Telomere, dass die Chromosomen mit jeder Zellteilung kürzer werden und umgeht so das End-Replikationsproblem.
Dieses Problem entsteht, da die DNA-Polymerase δ am diskontinuierlichen DNA-Strang ebenfalls von 5' nach 3' repliziert. Am diskontinuierlichen Strang synthetisiert die DNA-Polymerase δ ausgehend vom RNA-Primer die Okazaki-Fragmente, bis sie auf einen bestehenden RNA-Primer stößt. Durch Exonukleaseaktivität wird die erste RNA-Base am 5'-Ende des Primers entfernt. Die vollständige Entfernung des RNA-Primers übernehmen sogenannte Flap-Endonukleasen. Die Lücken des diskontinuierlichen DNA-Strangs werden ebenfalls mit Hilfe der Polymerase δ geschlossen und mit Desoxynukleotiden gefüllt. Eine Ligase verknüpft die noch bestehenden Strangbrüche. Da am diskontinuierlichen Strang DNA nur so lange repliziert werden kann, solange die Bindung eines RNA-Primers möglich ist, bleibt minimal ein Stück der Länge des RNA-Primers übrig. Abhängig von der Bindung des letzten RNA-Primers kann dieses Stück deutlich länger sein (bis zu 100 Nukleotide). Somit gehen Sequenzinformationen an den DNA-Enden verloren und die Chromosomenlänge nimmt mit jeder Zellteilung und der damit verbundenen DNA-Replikation ab (siehe Zellseneszenz).
In den meisten normalen Zellen ist die Aktivität der Telomerase nicht nachweisbar. Aktiv ist die Telomerase nur bei einzelligen Organismen sowie in sich kontinuierlich teilenden Zellen, wie Knochenmarkszellen, Zellen der Keimbahn (siehe auch Keimzelle), Embryonalzellen (= embryonale Stammzellen), Stammzellen sowie bei bestimmten Arten von Immunzellen bei mehrzelligen Organismen.
Die Telomerase ist außerdem aktiv in Krebszellen und verhilft ihnen dadurch dazu, sich unendlich oft zu teilen und im Körper zu wuchern. Nicht entartete Zellen können sich nur einer bestimmten Anzahl von Zellteilungen unterziehen (sogenannte Hayflick-Grenze).
Einzelnachweise
- ↑ C. W. Greider, E. H. Blackburn: Identification of a specific telomere terminal transferase activity in Tetrahymena extracts. In: Cell. Band 43, 1985, S. 405–413, doi:10.1016/0092-8674(85)90170-9, PMID 3907856.
- ↑ G. Gavory, M. Farrow, S. Balasubramanian: Minimum length requirement of the alignment domain of human telomerase RNA to sustain catalytic activity in vitro. In: Nucleic Acids Res. Vol. 30, Issue 20, Oktober 2002, S. 4470–4480. PMID 12384594, PMC 137139 (freier Volltext), doi:10.1093/nar/gkf575
Literatur
- W. K. Purves, J. Markl: Biologie. 7. Auflage. Elsevier, Spektrum Akad. Verlag, München 2006, ISBN 3-8274-1630-2.
Weblinks
- Umfangreiche Telomerase-Datenbank (englisch)
