Aminokwasy białkowe
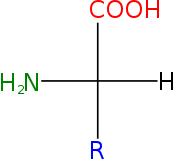
Aminokwasy białkowe – aminokwasy wchodzące w skład białek, łączące się z sobą wiązaniem peptydowym. Ze względu na pozycję grupy aminowej względem karboksylowej należą do tak zwanych α-aminokwasów, w których obie te grupy połączone są z atomem węgla α cząsteczki aminokwasu. Z wyjątkiem achiralnej glicyny, są to związki optycznie czynne o konfiguracji L, czyli mające grupę aminową po lewej stronie łańcucha głównego w projekcji Fischera.
Zazwyczaj wymienia się 20 podstawowych (tak zwanych kanonicznych) aminokwasów białkowych[1] kodowanych przez kodony. Niektóre źródła podają dodatkowo: selenometioninę[2], selenocysteinę oraz pirolizynę[3]. Selenocysteina ulega kotranslacyjnemu wbudowaniu w powstające białko, więc jest aminokwasem niekanonicznym, który nie powstaje poprzez modyfikacje potranslacyjne. W wyniku modyfikacji łańcuchów bocznych w trakcie translacji powstają: cystyna, hydroksylizyna, hydroksyprolina[4]. Istnieje kilka teorii ogólnych wyjaśniających czemu akurat te 20 konkretne aminokwasy zostały wybrane na drodze ewolucji[5][6][7]. W przypadku kilku aminokwasów znane są dokładne przyczyny np. ornityna i homoseryna ulegają cyklizacji zaburzając strukturę łańcucha głównego co skutkuje powstaniem białek o krótkim okresie półtrwania[8]. Inną przyczyną może być wysokie podobieństwo strukturalne niektórych aminokwasów przez co mogą być one omyłkowo włączone do białek np. kanawanina będąca bliskim analogiem argininy jest z tego powodu wysoce toksyczna[9].
Ze względu na zdolność organizmu do syntezy danego związku wyróżnia się aminokwasy endogenne i egzogenne.
D-Aminokwasy
Mimo że w przyrodzie dominują L-aminokwasy, spotyka się także ich enancjomery o konfiguracji D. Występują one naturalnie, na przykład w mikroorganizmach, roślinach i bezkręgowcach morskich. Powstają także podczas obróbki żywności, w wyniku racemizacji L-aminokwasów. Białka zawierające reszty D-aminokwasowe są gorzej trawione i mają niższą wartość odżywczą. Aktywność biologiczna D-aminokwasów różni się od naturalnych enancjomerów L, przy czym niektóre D-aminokwasy wywierają korzystny wpływ na organizm człowieka, a inne szkodliwy[10][11].
Podstawowe aminokwasy białkowe
Poniżej przedstawiono tabele zawierającą nazwy wraz ze skrótowymi oznaczeniami, podstawowe informacje oraz właściwości fizykochemiczne dla 20 standardowych aminokwasów.
| Skrót 1-liter. | Skrót 3-liter. | Nazwa | Wzór strukturalny | Polarność łańcucha bocznego | Charakter chemiczny[2] | Możliwość biosyntezy[12] (u człowieka) |
Budowa łańcucha bocznego |
Masa cząst.* | Punkt izoel. | pKa (α-COOH) |
pKb (α-+NH3) |
Promień van der Waalsa (ų) |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | Ala | alanina | 
|
niepolarny | obojętny | endogenny | alifatyczny | 89,09404 | 6,11 | 2,46 | 9,41 | 67 |
| C | Cys | cysteina | 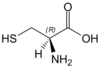
|
polarny | obojętny | endogenny | S | 121,15404 | 5,05 | 1,92 | 10,7 | 86 |
| D | Asp | kwas asparaginowy | 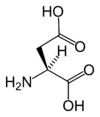
|
polarny | kwaśny | endogenny | COOH | 133,10384 | 2,85 | 1,99 | 9,9 | 91 |
| E | Glu | kwas glutaminowy | polarny | kwaśny | endogenny | COOH | 147,13074 | 3,15 | 2,10 | 9,47 | 109 | |
| F | Phe | fenyloalanina | 
|
niepolarny | obojętny | egzogenny | aromatyczny | 165,19074 | 5,49 | 2,2 | 9,31 | 135 |
| G | Gly | glicyna | 
|
niepolarny | obojętny | endogenny | H | 75,06714 | 6,06 | 2,35 | 9,78 | 48 |
| H | His | histydyna | 
|
polarny | zasadowy | egzogenny warunkowo | heterocykliczny | 155,15634 | 7,60 | 1,80 | 9,33 | 108 |
| I | Ile | izoleucyna | 
|
niepolarny | obojętny | egzogenny | alifatyczny | 131,17464 | 6,05 | 2,32 | 9,76 | 124 |
| K | Lys | lizyna | 
|
polarny | zasadowy | egzogenny | 146,18934 | 9,60 | 2,16 | 9,06 | 135 | |
| L | Leu | leucyna | 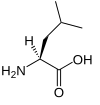
|
niepolarny | obojętny | egzogenny | alifatyczny | 131,17464 | 6,01 | 2,33 | 9,74 | 124 |
| M | Met | metionina | niepolarny | obojętny | egzogenny | S | 149,20784 | 5,74 | 2,13 | 9,28 | 124 | |
| N | Asn | asparagina | 
|
polarny | obojętny | endogenny | CONH2 | 132,11904 | 5,41 | 2,14 | 8,72 | 96 |
| P | Pro | prolina | 
|
niepolarny | obojetny | endogenny | heterocykliczny | 115,13194 | 6,30 | 1,95 | 10,64 | 90 |
| Q | Gln | glutamina | polarny | obojętny | endogenny | CONH2 | 146,14594 | 5,65 | 2,17 | 9,13 | 114 | |
| R | Arg | arginina | 
|
polarny | zasadowy | egzogenny warunkowo | 174,20274 | 10,76 | 1,82 | 8,99 | 148 | |
| S | Ser | seryna | 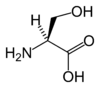
|
polarny | obojętny | endogenny | OH | 105,09344 | 5,68 | 2,19 | 9,21 | 73 |
| T | Thr | treonina | 
|
polarny | obojętny | egzogenny | OH | 119,12034 | 5,60 | 2,09 | 9,1 | 93 |
| V | Val | walina | 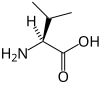
|
niepolarny | obojętny | egzogenny | alifatyczny | 117,14784 | 6,00 | 2,39 | 9,74 | 105 |
| W | Trp | tryptofan | niepolarny | obojętny | egzogenny | aromatyczny | 204,22844 | 5,89 | 2,46 | 9,41 | 163 | |
| Y | Tyr | tyrozyna | 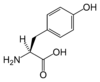
|
polarny | obojętny | endogenny | aromatyczny | 181,19124 | 5,64 | 2,20 | 9,21 | 141 |
* – masa cząsteczkowa na podstawie średniej ważonej uwzględniającej procentową zawartość izotopu danego pierwiastka w przyrodzie. Powstanie wiązania peptydowego powoduje zmniejszenie masy łańcucha polipeptydowego o jedną cząsteczkę wody (czyli o 18,01524 Da).
Ekspresja genetyczna
| Nazwa | Skrót 1-liter. | Skrót 3-liter. | Kodon(y) | Częstotliwość w
białkach Archeonów (%)& |
Częstotliwość w
białkach bakterii (%)& |
Częstotliwość w
białkach Eukaryontów (%)& |
Częstotliwość w
białkach człowieka |
Liczba cząsteczek
(×108) u (E. coli) |
|---|---|---|---|---|---|---|---|---|
| Alanina | A | Ala | GCU, GCC, GCA, GCG | 8,2 | 10,06 | 7,63 | 7,01 | 2,9 |
| Cysteina | C | Cys | UGU, UGC | 0,98 | 0,94 | 1,76 | 2,3 | 0,52 |
| Kwas asparaginowy | D | Asp | GAU, GAC | 6,21 | 5,59 | 5,4 | 4,73 | 1,4 |
| Kwas glutaminowy | E | Glu | GAA, GAG | 7,69 | 6,15 | 6,42 | 7,09 | 1,5 |
| Fenyloalanina | F | Phe | UUU, UUC | 3,86 | 3,89 | 3,87 | 3,65 | 1,1 |
| Glicyna | G | Gly | GGU, GGC, GGA, GGG | 7,58 | 7,76 | 6,33 | 6,58 | 3,5 |
| Histydyna | H | His | CAU, CAC | 1,77 | 2,06 | 2,44 | 2,63 | 0,54 |
| Izoleucyna | I | Ile | AUU, AUC, AUA | 7,03 | 5,89 | 5,1 | 4,33 | 1,7 |
| Lizyna | K | Lys | AAA, AAG | 5,27 | 4,68 | 5,64 | 5,72 | 2.0 |
| Leucyna | L | Leu | UUA, UUG, CUU, CUC, CUA, CUG | 9,31 | 10,09 | 9,29 | 9,97 | 2,6 |
| Metionina | M | Met | AUG | 2,35 | 2,38 | 2,25 | 2,13 | 0,88 |
| Asparagina | N | Asn | AAU, AAC | 3,68 | 3,58 | 4,28 | 3,58 | 1,4 |
| Pirolizyna | O | Pyl | UAG* | 0 | 0 | 0 | 0 | 0 |
| Prolina | P | Pro | CCU, CCC, CCA, CCG | 4,26 | 4,61 | 5,41 | 6,31 | 1,3 |
| Glutamina | Q | Gln | CAA, CAG | 2,38 | 3,58 | 4,21 | 4,77 | 1,5 |
| Arginina | R | Arg | CGU, CGC, CGA, CGG, AGA, AGG | 5,51 | 5,88 | 5,71 | 5,64 | 1,7 |
| Seryna | S | Ser | UCU, UCC, UCA, UCG, AGU, AGC | 6,17 | 5,85 | 8,34 | 8,33 | 1,2 |
| Treonina | T | Thr | ACU, ACC, ACA, ACG | 5,44 | 5,52 | 5,56 | 5,36 | 1,5 |
| Selenocysteina | U | Sec | UGA** | 0 | 0 | 0 | >0 | 0 |
| Walina | V | Val | GUU, GUC, GUA, GUG | 7,8 | 7,27 | 6,2 | 5,96 | 2,4 |
| Tryptofan | W | Trp | UGG | 1,03 | 1,27 | 1,24 | 1,22 | 0,33 |
| Tyrozyna | Y | Tyr | UAU, UAC | 3,35 | 2,94 | 2,87 | 2,66 | 0,79 |
| Kodon terminacyjny † | – | Term | UAA, UAG, UGA†† | – |
* UAG koduje normalnie kodon stopu (amber), jednak w przypadku niektórych bakterii i archeonów kodon ten skutkuje wstawieniem pirolizyny[13].
** UGA koduje normalnie kodon stopu (opal), jednak w przypadku niektórych bakterii i archeonów kodon ten skutkuje wstawieniem selenocysteiny[13].
† Kodony stopu zwykle nie kodują aminokwasów i zostały dodane dla kompletności kodu genetycznego.
†† Kodony UAG oraz UGA nie zawsze działają jako kodony stopu (patrz wyżej).
& Częstotliwość aminokwasów określono na podstawie średniej z proteomów (135 archeonów, 3775 bakterii, 614 eukariontów) oraz białek ludzkich (21006 białek)[14].
Produkcja przemysłowa
Roczna światowa produkcja podstawowych aminokwasów i ich soli wyniosła w 2005 r. ok. 3,3 mln ton, z czego ok. 95% stanowiła produkcja trzech z nich – kwasu glutaminowego i jego soli (głównie glutaminianu sodu), DL-metioniny i lizyny (głównie w postaci chlorowodorku)[15].
Zobacz też
Przypisy
- ↑ Lubert Stryer, Biochemia, wyd. 1, Warszawa: PWN, 1986, s. 505, ISBN 83-01-00140-2, wyd. 2, 2003, s. 765, ISBN 83-01-13978-1.
- ↑ a b Biologia: repetytorium dla maturzystów i kandydatów na wyższe uczelnie. Praca zbiorowa. Warszawa: Wydawnictwa Szkolne i Pedagogiczne, 2006, s. 15. ISBN 83-02-09004-2.
- ↑ Witold Mizerski: Tablice chemiczne. Warszawa: Wydawnictwo Adamantan, 2003, s. 280. ISBN 83-7350-031-6.
- ↑ Marzena Popielarska-Konieczna: Słownik szkolny. Biologia. Kraków: Wydawnictwo Zielona Sowa, 2003, s. 18. ISBN 83-7389-096-3.
- ↑ Albert Erives, A Model of Proto-Anti-Codon RNA Enzymes Requiring l-Amino Acid Homochirality, „Journal of Molecular Evolution”, 73 (1–2), 2011, s. 10–22, DOI: 10.1007/s00239-011-9453-4, PMID: 21779963, PMCID: PMC3223571 [dostęp 2023-01-20] (ang.).
- ↑ Michael Yarus, Jeremy Joseph Widmann, Rob Knight, RNA-amino acid binding: a stereochemical era for the genetic code, „Journal of Molecular Evolution”, 69 (5), 2009, s. 406–429, DOI: 10.1007/s00239-009-9270-1, PMID: 19795157 (ang.).
- ↑ Andrei S Rodin, Eörs Szathmáry, Sergei N Rodin, On origin of genetic code and tRNA before translation, „Biology Direct”, 6 (1), 2011, s. 14, DOI: 10.1186/1745-6150-6-14, PMID: 21342520, PMCID: PMC3050877 [dostęp 2023-01-20] (ang.).
- ↑ A.L. Weber, S.L. Miller, Reasons for the occurrence of the twenty coded protein amino acids, „Journal of Molecular Evolution”, 17 (5), 1981, s. 273–284, DOI: 10.1007/BF01795749, PMID: 7277510 [dostęp 2023-01-20] (ang.).
- ↑ G.A. Rosenthal, The biological effects and mode of action of L-canavanine, a structural analogue of L-arginine, „The Quarterly Review of Biology”, 52 (2), 1977, s. 155–178, DOI: 10.1086/409853, PMID: 331385 [dostęp 2023-01-20] (ang.).
- ↑ M. Friedman, Chemistry, nutrition, and microbiology of D-amino acids, „Journal of Agricultural and Food Chemistry”, 47 (9), 1999, s. 3457–3479, DOI: 10.1021/jf990080u, PMID: 10552672 (ang.).
- ↑ Mendel Friedman, Origin, microbiology, nutrition, and pharmacology of D-amino acids, „Chemistry & Biodiversity”, 7 (6), 2010, s. 1491–1530, DOI: 10.1002/cbdv.200900225, PMID: 20564567 [dostęp 2023-01-20] (ang.).
- ↑ Tablice biologiczne. Praca zbiorowa. Warszawa: Wydawnictwo Adamantan, 2003, s. 24. ISBN 83-7350-029-4.
- ↑ a b Michael Rother, Joseph A. Krzycki, Selenocysteine, pyrrolysine, and the unique energy metabolism of methanogenic archaea, „Archaea (Vancouver, B.C.)”, 2010, 2010, DOI: 10.1155/2010/453642, ISSN 1472-3654, PMID: 20847933, PMCID: PMC2933860 [dostęp 2018-06-15].
- ↑ Lukasz P. Kozlowski, Proteome-pI: proteome isoelectric point database, „Nucleic Acids Research”, 45 (D1), 2017, D1112–D1116, DOI: 10.1093/nar/gkw978, ISSN 1362-4962, PMID: 27789699, PMCID: PMC5210655 [dostęp 2018-06-15].
- ↑ Karlheinz Drauz i inni, Amino Acids, [w:] Ullmann’s Encyclopedia of Industrial Chemistry, Weinheim: Wiley‐VCH, 2006, s. 48, DOI: 10.1002/14356007.a02_057 (ang.).
